東アジア人の遺伝的歴史
Genetic history of East Asians

☆ この記事では、東アジアの人々の遺伝的特徴と人口動態の歴史(Genetic history of East Asians)、および東南アジアや北アジア、さらにはオセアニアの人々、そして一部、中央アジア、南アジア、ネイティブアメリカンといった遺伝的に関連 する集団とのつながりをまとめている。これらの集団は、集団遺伝学では「東ユーラシア人」と総称される。
| This
article summarizes the genetic makeup and population history of East
Asian peoples and their connection to genetically related populations
such as Southeast Asians and North Asians, as well as Oceanians, and
partly, Central Asians, South Asians, and Native Americans. They are
collectively referred to as "East Eurasians" in population genomics. https://en.wikipedia.org/wiki/Genetic_history_of_East_Asians |
こ
の記事では、東アジアの人々の遺伝的特徴と人口動態の歴史、および東南アジアや北アジア、さらにはオセアニアの人々、そして一部、中央アジア、南アジア、
ネイティブアメリカンといった遺伝的に関連する集団とのつながりをまとめている。これらの集団は、集団遺伝学では「東ユーラシア人」と総称される。 |
Overview Phylogenetic position of East Asian lineages among other Eastern Eurasians 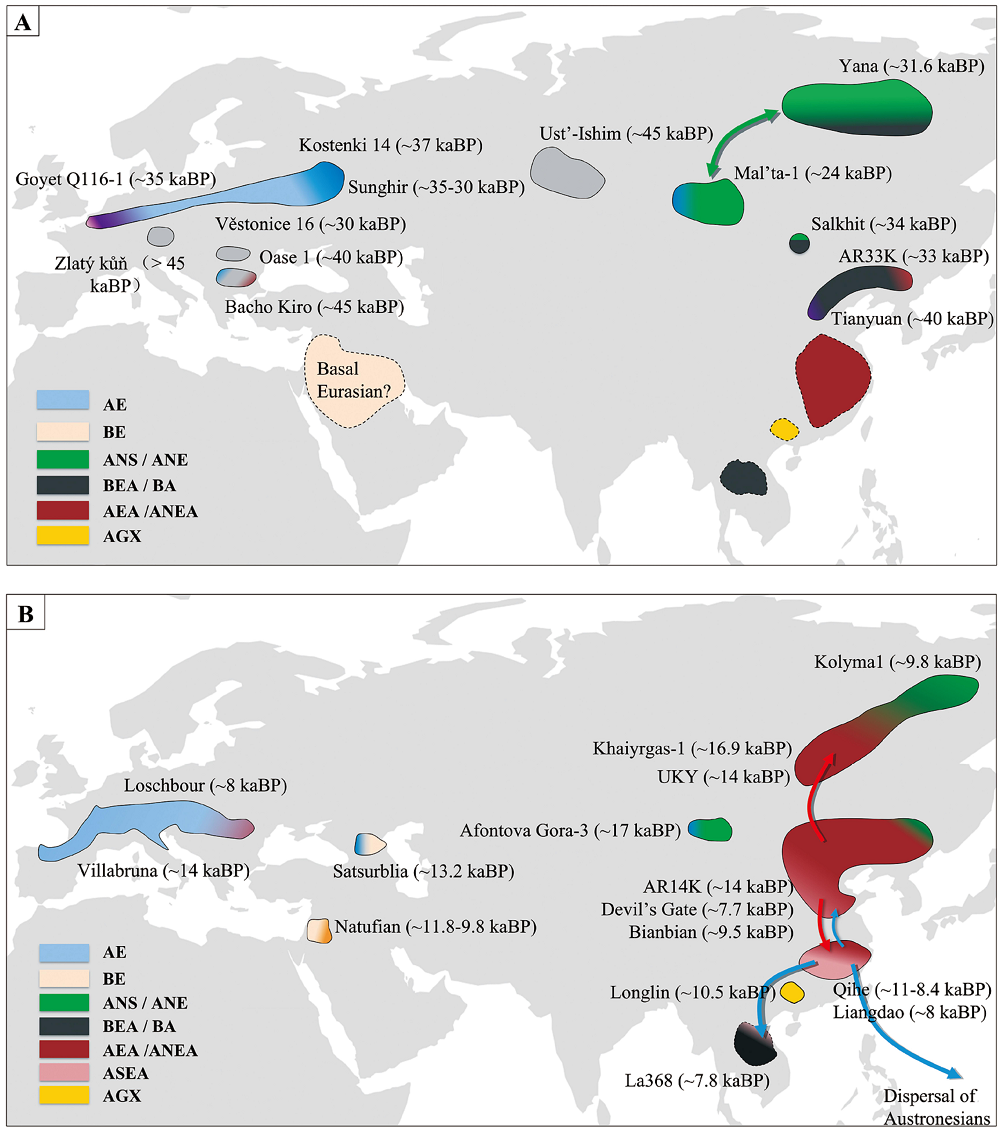 Schematic of Populations in Eurasia from 45 to 10 kaBP 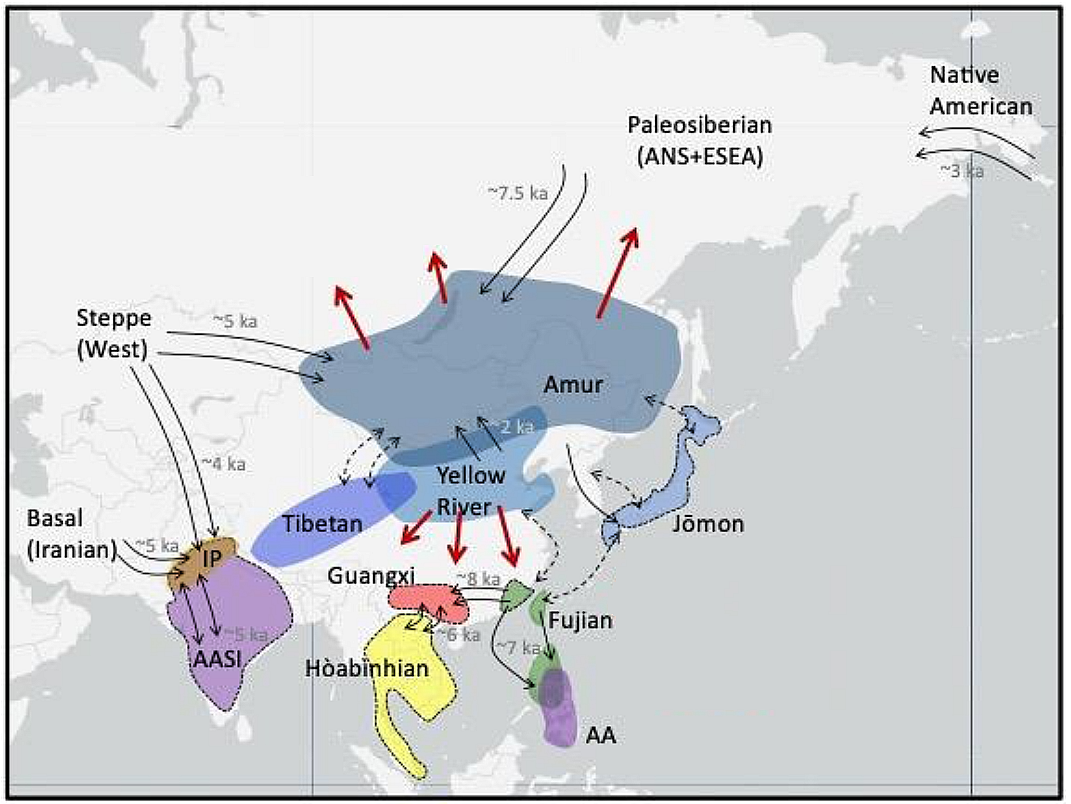 Highlighted regions show where ancient individuals associated with the labeled ancestry have been sampled. Population genomic research has studied the origin and formation of modern East Asians. The ancestors of East Asians (Ancient East Eurasians) split from other human populations possibly as early as 70,000 to 50,000 years ago. Possible routes into East Asia include a northern route model from Central Asia, beginning north of the Himalayas, and a southern route model, beginning south of the Himalayas and moving through Southeast Asia.[1][2] Seguin-Orlando et al. (2014) stated that East Asians diverged from West Eurasians, which occurred more than 36, 200 years ago in the Upper Paleolithic.[3] This divergence most likely occurred in the Persian Plateau.[4] Phylogenetic data suggests that an early Initial Upper Paleolithic wave (>45kya) "ascribed to a population movement with uniform genetic features and material culture" (Ancient East Eurasians) used a Southern dispersal route through South Asia, where they subsequently diverged rapidly, and gave rise to Australasians (Oceanians), the Ancient Ancestral South Indians (AASI), as well as Andamanese and East/Southeast Asians,[5] although Papuans may have also received some geneflow from an earlier group (xOoA),[6] around 2%,[7] next to additional archaic admixture in the Sahul region. The southern route model for East Asians has been corroborated in multiple recent studies, showing that most of the ancestry of Eastern Asians arrived from the southern route in to Southeast Asia at a very early period, starting perhaps as early as 70,000 years ago, and dispersed northward across Eastern Asia.[8][9][10][11][12][13] However, genetic evidence also supports more recent migrations to East Asia from Central Asia and West Eurasia along the northern route, as shown by the presence of haplogroups Q and R, as well as Ancient North Eurasian ancestry.[14][15] The southern migration wave likely diversified after settling within East Asia, while the northern wave, which probably arrived from the Eurasian steppe, mixed with the southern wave, probably in Siberia.[16] A review paper by Melinda A. Yang (in 2022) described the 'East- and Southeast Asian' lineage (ESEA); which is ancestral to modern East Asians, Southeast Asians, Polynesians, and Siberians, originated in Mainland Southeast Asia at c. 50,000 BCE, and expanded through multiple migration waves southwards and northwards, respectively. The ESEA lineage is also ancestral to the "basal Asian" Hoabinhian hunter-gatherers of Southeast Asia and the c. 40,000-year-old Tianyuan lineage found in Northern China, which can already be differentiated from the deeply related Ancestral Ancient South Indians (AASI) and Australasian (AA) lineages.[5] There are currently eight detected, closely related, sub-ancestries in the ESEA lineage: Amur ancestry (ANA) – Associated with populations in the Amur River region, Mongolia, and Siberia, as well as parts of Central Asia. Fujian ancestry – Associated with ancient samples in the Fujian region of Southern China, and modern Austronesian-speaking populations. Guangxi ancestry – Associated with a 10,500-year-old individual from Longlin, Guangxi. Jōmon ancestry – Ancestry associated with 8,000–3,000-year-old individuals in the Japanese archipelago. Hoabinhian ancestry – Ancestry on the ESEA lineage associated with 8,000–4,000-year-old hunter-gatherers in Laos and Malaysia. Tianyuan ancestry – Ancestry on the ESEA lineage associated with an Upper Paleolithic individual dating to 40,000 years ago in northern China. Tibetan ancestry – Associated with 3,000-year-old individuals in the Himalayan region of the Tibetan Plateau. Yellow River ancestry – Associated with populations in the Yellow River region and common among Sino-Tibetan-speakers. 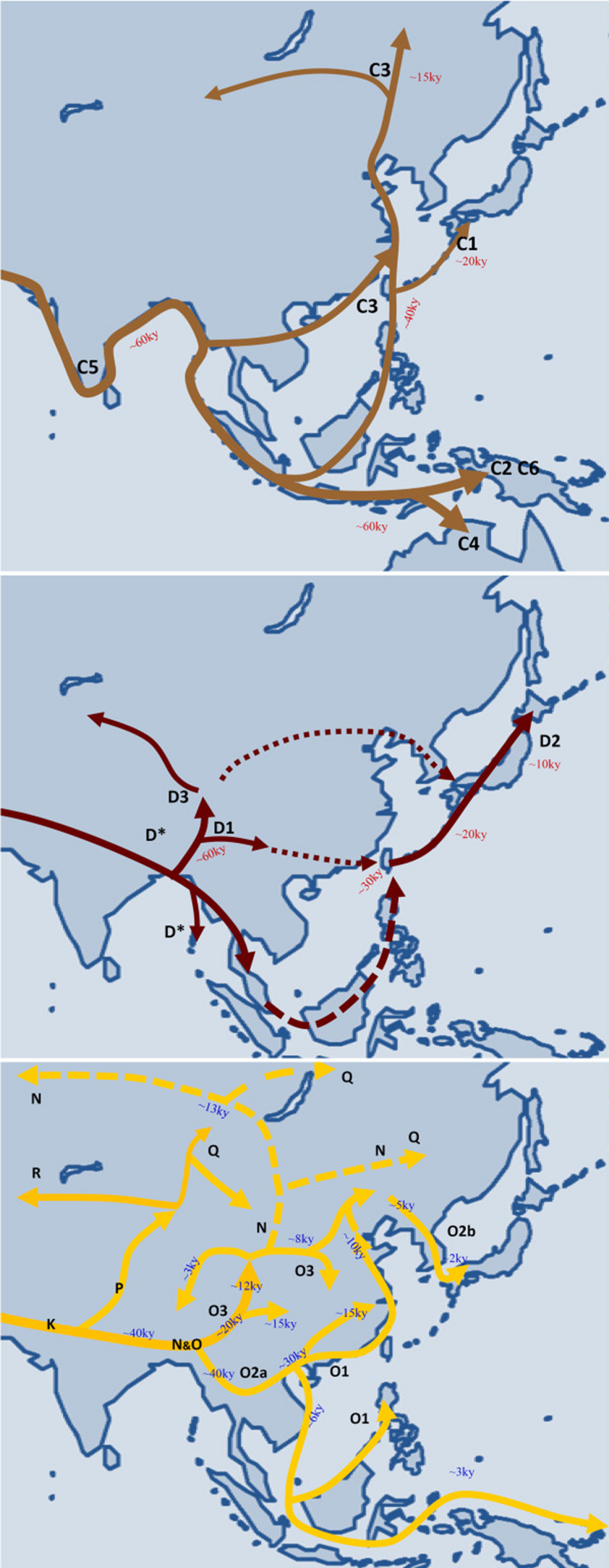 Proposed migration routes (Wang 2013) of dominant East Asian paternal haplogroups (C, D, N, and O), during the peopling of East Asia[17] Modern Northeast Asians derive most of their ancestry from the "Amur" (Ancient Northeast Asian) sub-linesge, which expanded massively with millet cultivation and pastoralism. Modern Southeast Asians (specifically Austronesians) mainly carry "Fujian" (Ancient Southern East Asian) ancestry, which is associated with the spread of rice cultivation. Contemporary East Asians (most notably Sino-Tibetan speakers) mostly have Yellow River ancestry, associated with both millet and rice cultivation. "East Asian Highlanders" (Tibetans) carry both Tibetan ancestry and Yellow River ancestry. Japanese people were found to have a tripartite origin; consisting of Jōmon ancestry, Amur ancestry, and Yellow River ancestry. Indigenous peoples of the Americas formed from Ancient North Eurasians and from an early Northern East Asian branch, giving rise to "Ancient Paleo-Siberians", which in turn gave rise to both "modern Paleosiberians" and contemporary Native Americans. Isolated hunter-gatherers in Southeast Asia, specifically in Malaysia and Thailand, such as the Semang, derive most of their ancestry from the Hoabinhian lineage.[18][19][20][21][22][23][24]  Proposed migration routes of maternal haplogroups during the peopling of Eurasia[25] The genetic makeup of East Asians is primarily characterized by the Ancient Northern East Asian (ANEA) and Ancient Southern East Asian (ASEA) lineages, which diverged from each other at least 19,000 years ago, after the divergence of the Jōmon, Longlin, Hoabinhian and Tianyuan lineages.[26][5] East Asian populations exhibit some European admixture, originating from Silk Road traders and interactions with Mongolians, who were well-acquainted with European-like populations. This is more common among northern Han Chinese (2.8%) than southern Han Chinese (1.7%), Japanese (2.2%), and Koreans (1.6%). Although East Asians have less European-like admixture than Northeast Asians like Mongolians (10.9%), Oroqen (9.6%), Daur (8.0%), and Hezhen (6.8%).[27] East Asians carry a variation of the MFSD12 gene, which is responsible for lighter skin colour.[28] Huang et al. (2021) additionally found evidence for light skin being selected among the ancestral populations of Europeans and East Asians, prior to their divergence.[29] |
概要 東アジアの系統が他の東アジア系の人々の中で占める系統発生上の位置  45,000~10,000年前のユーラシア大陸の集団の概略図  ハイライトされた地域は、標識された祖先を持つ古代の個体が採取された場所を示す。 集団ゲノム研究では、現代の東アジア人の起源と形成が研究されている。東アジア人の祖先(古代東ユーラシア人)は、おそらく7万~5万年前には他の人類集 団から分岐していた。東アジアへの進出経路としては、ヒマラヤの北側から中央アジアを経由する北ルートモデルと、ヒマラヤの南側から東南アジアを通過する 南ルートモデルが考えられる。[1][2] Seguin-Orlando et al. ( 2014年)は、東アジア人と西ユーラシア人が分岐したのは、3万6200年以上前の旧石器時代後期の上部旧石器時代であったと述べている。[3] この分岐は、おそらくペルシア高原で起こったと考えられている。[4] 系統発生学的データによると、初期の旧石器時代前期(45,000年前以上)の波は、「均一な遺伝的特徴と物質文化を持つ人口移動に起因する」(古代東 ユーラシア人)もので、南アジアを通る南の分散ルートを使用し、その後急速に分化し、 古代南インド人(AASI)やアンダマン人、東・東南アジア人も含むが、パプア人もまた、サフル地域における古い混血に加えて、2%ほど、より初期のグ ループ(xOoA)からの遺伝子流入を受けている可能性がある。 東アジア人の南方ルートモデルは、複数の最近の研究で裏付けられており、東アジア人の祖先のほとんどは、7万年前という非常に早い時期に東南アジアに南方 ルートから到達し、東アジア北部に分散したことが示されている。 。しかし、ハプログループQとRの存在や古代北ユーラシア人の祖先が示すように、中央アジアや西ユーラシアから北のルートを通って東アジアへ移動したとい う最近の証拠も遺伝学的な証拠によって裏付けられている。 南からの移住の波は、東アジアに定住した後、おそらく多様化した。一方、ユーラシア大草原からやってきたと思われる北からの波は、おそらくシベリアで南からの波と混ざり合った。 メリンダ・A・ヤン(2022年)による総説論文では、「東・東南アジア系」系統(ESEA)について説明している。これは、現代の東アジア人、東南アジ ア人、ポリネシア人、シベリア人の共通祖先であり、およそ5万年前に東南アジア大陸部で誕生し、複数の移住波によってそれぞれ南と北に拡大した。ESEA 系統は、東南アジアの「基底アジア人」であるホアビン文化の狩猟採集民や、中国北部で発見された約4万年前の天元系統の祖先でもある。これらは、古代南イ ンド人(AASI)やオーストラレーシア人(AA)の系統と深く関連しているが、すでに区別することができる。[5] ESEA系統には、現在、密接に関連した8つの祖先系統が検出されている。 アムール先祖(ANA) – アムール川流域、モンゴル、シベリア、および中央アジアの一部の集団に関連している。 福建省先祖 – 中国南部の福建省地域における古代のサンプル、および現代のオーストロネシア語族話者集団に関連している。 広西の祖先 – 広西の龍林から出土した1万5000年前の個体に関連する。 縄文人の祖先 – 日本列島における8000~3000年前の個体に関連する。 ホアビン人の祖先 – ラオスとマレーシアにおける8000~4000年前の狩猟採集民に関連するESEA系統の祖先。 天元系祖先 – ESEA系統の祖先で、中国北部で4万年前の旧石器時代後期の人類と関連している。 チベット系祖先 – チベット高原のヒマラヤ地域で3000年前の人類と関連している。 黄河系祖先 – 黄河流域の集団と関連しており、シナ・チベット語話者に多く見られる。  東アジアの主要な父系ハプログループ(C、D、N、O)の移住ルート(Wang 2013)の提案。東アジアへの人類の拡散期[17] 現代の北東アジア人は、その祖先のほとんどを「アムール」(古代北東アジア)の支系統に由来しており、この系統はアワ栽培と牧畜によって大規模に拡大し た。現代の東南アジア人(特にオーストロネシア人)は主に「福建」(古代東南アジア)の系統を継承しており、これは稲作の広がりと関連している。現代の東 アジア人(特にシナ・チベット語族)は主に黄河の系統を継承しており、これはアワ栽培と稲作の両方に関連している。「東アジア高地民族」(チベット人) は、チベット系と黄河系の両方の祖先を持つ。日本人は、縄文系、アムール系、黄河系の3つの起源を持つことが分かっている。アメリカ大陸の先住民は、古代 北ユーラシア人と初期の北東アジアの分派から形成され、「古代パレオ・シベリア人」を生み出し、さらに「現代パレオ・シベリア人」と現代のネイティブアメ リカンを生み出した。東南アジア、特にマレーシアやタイの孤立した狩猟採集民であるセマン族は、その祖先のほとんどをホアビン人の系統に由来している。 [18][19][20][21][22][23][24]  ユーラシアへの人類拡散における母系ハプログループの移動ルート(推定)[25] 東アジア人の遺伝的構成は、主に古代北東アジア(ANEA)と古代南東アジア(ASEA)の系統によって特徴づけられる。これらの系統は、縄文、龍仁、ホアビン、天元の各系統が分岐した後、少なくとも1万9000年前に分岐したものである。[26][5] 東アジアの集団には、ヨーロッパ人との混血が一部見られ、その起源はシルクロードの商人や、ヨーロッパ人によく似た人々をよく知っていたモンゴル人との交 流にある。これは、北方漢族(2.8%)よりも南方漢族(1.7%)、日本人(2.2%)、韓国人(1.6%)の間でより一般的である。東アジア人は、モ ンゴル人(10.9%)、オロチョン人(9.6%)、ダウール人(8.0%)、ヘゼン人(6.8%)のような北東アジア人よりもヨーロッパ人との混血が少 ないが、[27] 東アジア人は、肌の色を薄くするMFSD12遺伝子の変異体を持っている。[28] 黄ら(2021年)はさらに、ヨーロッパ人と東アジア人の祖先集団において、分岐前に肌の色が薄い個体が選択されていた証拠を発見した。[29] |
| Ancient and historical populations Xiongnu people Main article: Xiongnu The Xiongnu, possibly a Turkic, Mongolic, Yeniseian or multi-ethnic people, were a confederation[30] of nomadic peoples who, according to ancient Chinese sources, inhabited the eastern Eurasian Steppe from the 3rd century BC to the late 1st century AD. Chinese sources report that Modu Chanyu, the supreme leader after 209 BC, founded the Xiongnu Empire.[31] Autosomal DNA It was found that the "predominant part of the Xiongnu population is likely to have spoken Turkic". However, important cultural, technological and political elements may have been transmitted by Eastern Iranian-speaking Steppe nomads: "Arguably, these Iranian-speaking groups were assimilated over time by the predominant Turkic-speaking part of the Xiongnu population".[32] This is reflected by the average genetic makeup of Xiongnu samples, having approximately 58% East Eurasian ancestry, represented by a Bronze Age population from Khövsgöl, Mongolia, which may be associated with the Turkic linguistic heritage. The rest of the Xiongnu's ancestry (~40%) was related to West Eurasians, represented by the Gonur Depe BMAC population of Central Asia, and the Sintashta culture of the Western steppe.[32][33] The Xiongnu displayed striking heterogeneity and could be differentiated into two subgroups, "Western Xiongnu" and "Eastern Xiongnu", with the former being of "hybrid" origins displaying affinity to previous Saka tribes, such as represented by the Chandman culture, while the later was of primarily Ancient Northeast Asian (Ulaanzuukh-Slab Grave) origin.[34][32] High status Xiongnu individuals tended to have less genetic diversity, and their ancestry was essentially derived from the Eastern Eurasian Ulaanzuukh/Slab Grave culture.[35] Paternal lineages A review of the available research has shown that, as a whole, 53% of Xiongnu paternal haplogroups were East Eurasian, while 47% were West Eurasian.[36] In 2012, Chinese researchers published an analysis of the paternal haplogroups of 12 elite Xiongnu male specimens from Heigouliang in Xinjiang, China. Six of the specimens belonged to Q1a, while four belonged to Q1b-M378. 2 belonged to unidentified clades of Q*.[37] In another study, a probable Chanyu of the Xiongnu empire was assigned to haplogroup R1.[38][39] Maternal lineages The bulk of the genetics research indicates that, as a whole, 73% of Xiongnu maternal haplogroups were East Eurasian, while 27% were West Eurasian.[40] A 2003 study found that 89% of Xiongnu maternal lineages from the Egiin Gol valley were of East Asian origin, while 11% were of West Eurasian origin.[41] A 2016 study of Xiongnu from central Mongolia found a considerably higher frequency of West Eurasian maternal lineages, at 37.5%.[42] Xianbei people Main article: Xianbei Autosomal DNA A full genome study on multiple Xianbei remains found them to be derived primarily to exclusively from the Ancient Northeast Asian gene pool.[43] Paternal lineages A genetic study published in the American Journal of Physical Anthropology in August 2018 noted that the paternal haplogroup C2b1a1b has been detected among the Xianbei and the Rouran, and was probably an important lineage among the Donghu people.[44] Maternal lineages Genetic studies published in 2006 and 2015 revealed that the mitochondrial haplogroups of Xianbei remains were of East Asian origin. According to Zhou (2006) the maternal haplogroup frequencies of the Tuoba Xianbei were 43.75% haplogroup D, 31.25% haplogroup C, 12.5% haplogroup B, 6.25% haplogroup A and 6.25% "other".[45] Zhou (2014) obtained mitochondrial DNA analysis from 17 Tuoba Xianbei, which indicated that these specimens were, similarly, completely East Asian in their maternal origins, belonging to haplogroups D, C, B, A, O and haplogroup G.[46][47] Jōmon people Main article: Jōmon people The Jōmon people represent the indigenous population of the Japanese archipelago during the Jōmon period. They are inferred to descend from the Paleolithic inhabitants of Japan. Genetic analyses on Jōmon remains found them to represent a deeply diverged East Asian lineage. The Jōmon lineage is inferred to have diverged from Ancient East Asians before the divergence between Ancient Northern East Asians and Ancient Southern East Asians, but after the divergence of the basal Tianyuan man and or Hoabinhians. Beyond their broad affinity with Eastern Asian lineages, the Jōmon also display a weak affinity for Ancient North Eurasians (ANE), which may be associated with the introduction of microblade technology to Northeast Asia and northern East Asia during the Last Glacial Maximum via the ANE or Ancient Paleo-Siberians.[48][49] Hoabinhians Main article: Hoabinhian The Hoabinhians represent a technologically advanced society of hunter-gatherers, primarily living in Mainland Southeast Asia, but also adjacent regions of Southern China. While the Upper Paleolithic origins of this 'Hoabinhian ancestry' are unknown, Hoabinhian ancestry has been found to be related to the main 'East Asian' ancestry component found in most modern East and Southeast Asians, although deeply diverged from it.[50][51] Together with the Paleolithic Tianyuan man, they form early branches of East Asian genetic diversity, and are described as "Basal Asian" (BA) or "Basal East Asian" (BEA).[52] |
古代および歴史的な民族 匈奴 詳細は「匈奴」を参照 匈奴は、おそらくテュルク系、モンゴル系、エニセイ人、あるいは多民族の集団であり、古代中国の資料によると、紀元前3世紀から紀元後1世紀後半にかけて ユーラシア大陸東部の草原地帯に居住していた遊牧民族の連合国家であった。中国の資料によると、紀元前209年以降の最高指導者であるモドゥ・チャヌが匈 奴帝国を建国したとされる。 常染色体DNA 匈奴の人口の大半がテュルク語を話していた可能性が高いことが判明した。しかし、重要な文化、技術、政治的要素は、東イラン語を話す草原遊牧民によって伝 えられた可能性がある。「おそらく、これらのイラン語話者集団は、匈奴の人口の大部分を占めるテュルク語話者集団に、長い時間をかけて同化していった」の である。[32] これは、モンゴルのフブスゴルから出土した青銅器時代の人口に代表される、約58%の東ユーラシア系祖先を持つ匈奴サンプルの平均的な遺伝子構成に反映さ れている。この遺伝子構成は、テュルク語の言語的遺産と関連している可能性がある。匈奴の残りの祖先(約40%)は西ユーラシア人と関連しており、中央ア ジアのゴヌル・デペのBMAC民族や西草原のシンシュタ文化に代表される。[32][33] 匈奴は著しい異質性を示し、2つのサブグループ、「西匈奴」と「東匈奴」に区別することができ、前者は 「ハイブリッド」起源で、チャンドマン文化に代表されるような以前のサカ族と親和性がある一方、後者は主に古代北東アジア(ウランズーフ・スラブ墓)起源 であった。[34][32] 地位の高い匈奴の個人は遺伝的多様性が少ない傾向があり、彼らの祖先は基本的に東ユーラシアのウランズーフ・スラブ墓文化に由来していた。[35] 父系 利用可能な研究のレビューでは、全体として、匈奴の父系ハプログループの53%が東ユーラシア系、47%が西ユーラシア系であることが示されている。 [36] 2012年、中国の研究者らは、中国・新疆の黒谷良から出土した12体のエリート匈奴男性標本の父系ハプログループの分析結果を発表した。標本の6つは Q1aに属し、4つはQ1b-M378に属した。2つは未同定のQ*クレードに属した。[37] 別の研究では、匈奴帝国のチャヌ(単于)と思われる人物はハプログループR1に分類された。[38][39] 母系 大部分の遺伝学的研究は、全体として、匈奴の母系ハプログループの73%が東ユーラシア系であり、27%が西ユーラシア系であることを示している。 [40] 2003年の研究では、 エジンゴル渓谷の89%が東アジア起源であり、11%が西ユーラシア起源であった。[41] 2016年にモンゴル中央部の匈奴を対象とした研究では、西ユーラシアの母系家系が37.5%と、かなり高い頻度で発見された。[42] 鮮卑 詳細は「鮮卑」を参照 常染色体DNA 複数の鮮卑の遺骨に関する全ゲノム研究により、それらの遺骨は主に、あるいはもっぱら古代北東アジアの遺伝子プールに由来することが判明した。[43] 父系 2018年8月に『アメリカン・ジャーナル・オブ・フィジカル・アンソロポロジー』誌で発表された遺伝学の研究では、父系ハプログループC2b1a1bが鮮卑と楼蘭で検出されており、おそらく東胡の人々の中でも重要な系統であったことが指摘されている。[44] 母系 2006年と2015年に発表された遺伝学の研究により、鮮卑のミトコンドリアハプログループは東アジア起源であることが明らかになった。周(2006 年)によると、當波鮮卑の母系ハプログループの頻度は、ハプログループDが43.75%、ハプログループCが31.25%、ハプログループBが 12.5%、ハプログループAが6.25%、そして「その他」が6.2 5%「その他」であった。[45] 周(2014年)は、17人の吐跋鮮卑のミトコンドリアDNA分析を行い、これらの標本も同様に、母系が完全に東アジア起源であり、ハプログループD、 C、B、A、O、およびハプログループGに属することが示された。[46][47] 縄文人 詳細は「縄文人」を参照 縄文人は、縄文時代における日本列島の先住民族である。彼らは、日本の旧石器時代の住民の子孫であると推定されている。遺伝子解析により、縄文人の遺伝子 は、東アジアの系統の中でも特に分化したものであることが判明した。縄文人の系統は、古代東アジア人と古代北東アジア人、古代南東アジア人の分化以前に分 化したと推定されているが、基底集団である天元人の分化、あるいはホアビン人の分化以降である。東アジアの系統との広範な類縁関係に加え、縄文人は古代北 ユーラシア人(ANE)との弱い類縁関係も示しており、これは最終氷期最盛期にマイクロブレード技術が古代パレオ・シベリア人(ANE)または古代パレ オ・シベリア人を通じて北東アジアおよび東アジア北部に伝来したことと関連している可能性がある。[48][49] ホアビン人 詳細は「ホアビン文化」を参照 ホアビン人は、主に東南アジア大陸部に、また一部は中国南部の隣接地域に居住していた、狩猟採集民の中でも技術的に高度な社会を代表する。この「ホアビン 人の祖先」の旧石器時代における起源は不明であるが、ホアビン人の祖先は、現代の東アジア人と東南アジア人の大半に見られる主要な「東アジア人」の祖先構 成要素と関連していることが判明している。ただし、その構成要素とは大きく分岐している 。旧石器時代の天元人の人類とともに、彼らは東アジアの遺伝的多様性の初期の枝を形成しており、「基底アジア人(BA)」または「基底東アジア人 (BEA)」と表現されている。[52] |
Modern populations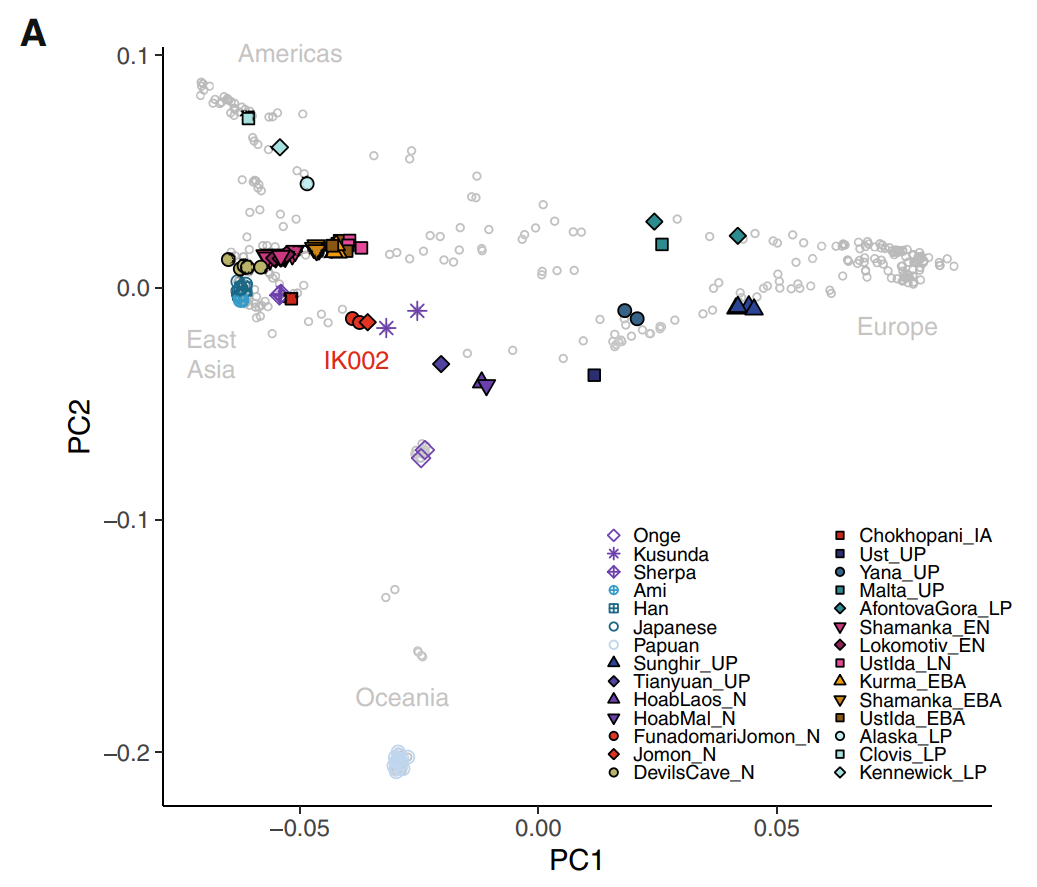 Genetic structure of present-day and ancient Eurasians[53] Main article: East Asian peoples Manchu and Daur peoples Main article: Tungusic peoples Autosomal DNA A study on the Manchu population of Liaoning reported that they have a close genetic relationship and significant admixture signals from northern Han Chinese. The Liaoning Manchu were formed from a major ancestral component related to Yellow River farmers and a minor ancestral component linked to ancient populations from the Amur River Basin, or others. The Manchu were therefore an exception to the coherent genetic structure of Tungusic-speaking populations, likely due to the large-scale population migrations and genetic admixtures in the past few hundred years.[54] Paternal lineages A plurality of Daur males belong to Haplogroup C-M217 (12/39 = 30.8% according to Xue Yali et al. 2006,[55] 88/207 = 42.5% according to Wang Chi-zao et al. 2018[56]), with Haplogroup O-M122 being the second most common haplogroup among present-day Daurs (10/39 = 25.6%,[55] 52/207 = 25.1%[56]). There are also tribes (hala; cf. Kazakh tribes) among the Daurs that belong predominantly to other Y-DNA haplogroups, such as Haplogroup N-M46/M178 (Merden hala) and Haplogroup O1b1a1a-M95 (Gobulo hala).[56] Haplogroup C3b2b1*-M401(xF5483)[57][58][59] has been identified as a possible marker of the Aisin Gioro and is found in ten different ethnic minorities in northern China, but is largely absent from Han Chinese.[60][61][59] The Manchu people also display a significant amount of haplogroup C-M217, but the most often observed Y-DNA haplogroup among present-day Manchus is Haplogroup O-M122, which they share in common with the general population of China.[62][55][63] Ainu people Main article: Ainu people The exact origins of the early Ainu remains unclear, but it is generally agreed to be linked to the Satsumon culture of the Epi-Jōmon period, with later influences from the nearby Okhotsk culture.[64] The Ainu appear genetically most closely related to the Jōmon period peoples of Japan. The genetic makeup of the Ainu represents a "deep branch of East Asian diversity". Compared to contemporary East Asian populations, the Ainu share "a closer genetic relationship with northeast Siberians".[8][49] Japanese people 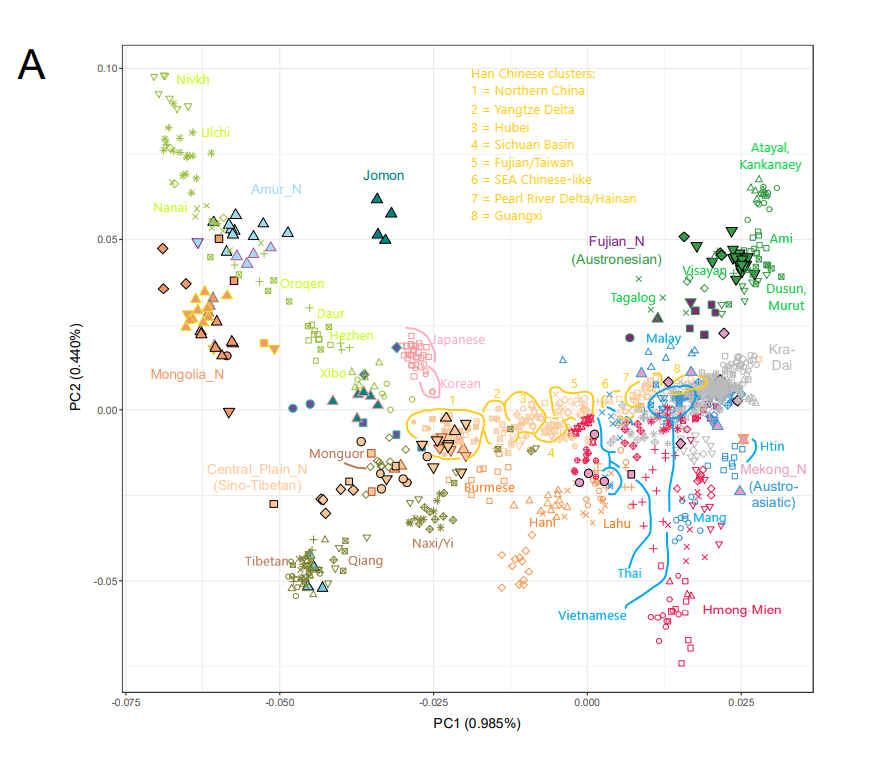 A population genomic PCA graph, showing the substructure of Eastern Asian populations Main articles: Japanese people and Genetic and anthropometric studies on Japanese people Japanese populations in modern Japan can be traced to three separate, but related demographics: the Ainu, Ryukyuan and Mainland Japanese (Yamato people). The populations are closely related to clusters found in North-Eastern Asia[65][66][67] with the Ainu group being most similar to Ryukyuans[66][68] and the Yamato group being most similar to Koreans[69][70][71] among other East Asian people. Autosomal DNA The majority of Japanese genetic ancestry is derived from sources related to other mainland Asian groups, mostly Koreans, while the other amount is derived from the local Jōmon hunter-gatherers.[71] 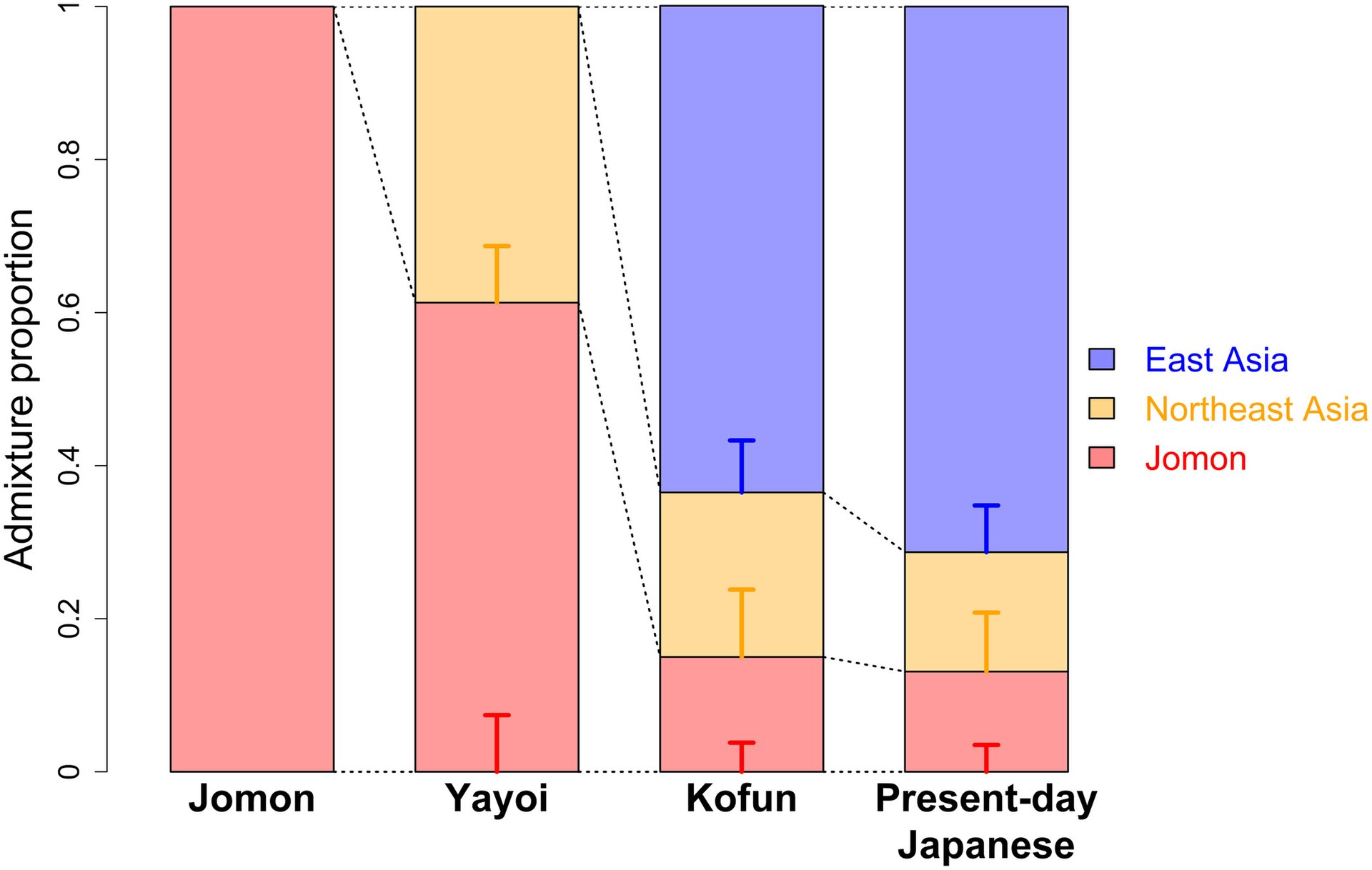 Geomic transitions in parallel with cultural transitions in pre- and protohistoric Japan According to a full genome analyses, the modern Japanese harbor a Northeast Asian, an East Asian, and an indigenous Jōmon component. In addition to the indigenous Jōmon hunter-gatherers and the Yayoi period migrants, a new strand was hypothesized to have been introduced during the Yayoi-Kofun transition period that had strong cultural and political affinity with Korea and China.[72] Paternal lineages A comprehensive study of worldwide Y-DNA diversity (Underhill et al. 2000) included a sample of 23 males from Japan, of whom 35% belonged to haplogroup D-M174, 26% belonged to O-M175, 22% belonged to O-M122, 13% belonged to C-M8 and C-M130, and 4.3% belonged to N-M128.[73] Poznik et al. (2016) reported the haplogroups of a sample of Japanese men from Tokyo:[74] 36% belonged to D2-M179, 32% had O2b-M176, 18% carried O3-M122, 7.1% carried C1a1-M8, 3.6% belonged to O2a-K18, and 3.6% carried C2-M217.[75] Maternal lineages According to an analysis of the 1000 Genomes Project's sample of Japanese collected in the Tokyo metropolitan area, the mtDNA haplogroups found among modern Japanese include D (35.6%), B (13.6%), M7 (10.2%), G (10.2%), N9 (8.5%), F (7.6%), A (6.8%), Z (3.4%), M9 (2.5%), and M8 (1.7%).[76] Korean people Main article: Koreans  Regional reference panel, PCA, and Admixture analysis Modern Koreans are overall more similar to northeast Asians than to southeast Asians.[77] Autosomal DNA Ancient genome comparisons revealed that the genetic makeup of Koreans can be best described as an admixture between Northeast Asian hunter-gatherers and an influx of rice-farming Southeast Asian agriculturalists from the Yangtze river valley.[77] This is supported by archaeological, historical and linguistic evidence, which suggest that the direct ancestors of Koreans were proto-Koreans who inhabited the northeastern region of China and the Korean Peninsula during the Neolithic (8,000–1,000 BC) and Bronze (1,500–400 BC) Ages.[78] There is evidence for considerable genetic diversity,[79] including elevated levels of Jōmon ancestry among early southern Koreans.[80] It was hypothesized that the Jōmon ancestry of ancient Koreans was lost over time, as they continually mixed with incoming populations from northern China,[81] followed by a period of isolation during the Three Kingdoms period, resulting in the homogenous gene pool of modern Koreans.[82][79] A 2022 study was unable to detect significant Jōmon ancestry in modern Koreans, however by using different proxies of ancestry, a Jōmon contribution of 3.1–4.4% was found for present-day Ulsan Koreans. Nevertheless, the authors suggested that the model that yielded this result is not the most reliable.[83] Evidence for both Southern and Northern mtDNA and Y-DNA haplogroups has been observed in Koreans, similar to Japanese.[53] Over 70% of extant genetic diversity among Koreans can be explained by admixture with ancient South Chinese immigrants, who were related to Iron Age Cambodians.[84] Paternal lineages Studies of polymorphisms in the human Y-chromosome have so far produced evidence to suggest that the Korean people have a long history as a distinct, mostly endogamous ethnic group, with successive waves of people moving to the peninsula and three major Y-chromosome haplogroups.[85] A majority of Koreans belong to subclades of haplogroup O-M175 (ca. 79% in total,[78][86] with about 42%[86] to 44%[78] belonging to haplogroup O2-M122, about 31%[78] to 32%[86] belonging to haplogroup O1b2-M176, and about 2%[78] to 3%[86] belonging to haplogroup O1a-M119), while a significant minority belong to subclades of haplogroup C2-M217 (ca. 12%[78] to 13%[86] in total). Other Y-DNA haplogroups, including haplogroup N-M231, haplogroup D-M55, and haplogroup Q-M242, are also found in smaller proportions of present-day Koreans.[87][88][89] Maternal lineages Studies of Korean mitochondrial DNA lineages have shown that there is a high frequency of Haplogroup D4, followed by haplogroup B, and then haplogroup A and haplogroup G. Haplogroups with lower frequency include N9, Y, F, D5, M7, M8, M9, M10, M11, R11, C, and Z.[90][91][92] Mongolic peoples Main article: Mongolic peoples The ethnogenesis of Mongolic peoples is largely linked with the expansion of Ancient Northeast Asians. They subsequently came into contact with other groups, notably Sinitic peoples to their South and Western Steppe Herders to their far West. The Mongolians pastoralist lifestyle, may in part be derived from the Western Steppe Herders, but without much geneflow between these two groups, suggesting cultural transmission.[93][94] The Mongols are believed to be the descendants of the Xianbei and the proto-Mongols. The former term includes the Mongols proper (also known as the Khalkha Mongols), Oirats, the Kalmyk people and the Southern Mongols. The latter comprises the Abaga Mongols, Abaganar, Aohans, Baarins, Gorlos Mongols, Jalaids, Jaruud, Khishigten, Khuuchid, Muumyangan and Onnigud. The Daur people are descendants of the para-Mongolic Khitan people.[95] Paternal lineages The majority of Mongols in Mongolia and Russia belong to subclades of haplogroup C-M217,[96] followed by lower frequency of O-M175 and N-M231.[97] A minority belongs to haplogroup Q-M242, and a variety of West Eurasian haplogroups.[98] Maternal lineages The maternal haplogroups are diverse but similar to other northern Asian populations, including Haplogroup D, Haplogroup C, Haplogroup B, and Haplogroup A, which are shared among indigenous American and Asian populations.[99] West Eurasian mtDNA haplogroups makes up a some minority percentages. Haplogroup HV, Haplogroup U, Haplogroup K, Haplogroup I, Haplogroup J are all found in Mongolic people.[100] Han Chinese See also: Han Chinese Han Chinese descend primarily from Neolithic Yellow River farmers, which formed primarily from Ancient Northern East Asians with some contributions from Ancient Southern East Asians. Northern Han Chinese mostly carry ANEA ancestry with a moderate degree of ASEA admixture, whereas southern Han Chinese carry significantly higher levels of ASEA ancestry than Northern Han, although ANEA ancestry still predominates.[101][26][102] The Han Chinese show a close genetic relationship with other modern East Asian populations such as the Koreans and Yamato.[103][104][105][106][107][108][109] A 2018 research paper found that while the Han Chinese are closely related to the Koreans and Yamato in terms of a correlative genetic relationship, they are also easily genetically distinguishable from them. And that the same Han Chinese subgroups are genetically closer to each other relative to their Korean and Yamato counterparts, but are still easily distinguishable from each other.[109] Research published in 2020 found the Yamato Japanese population to be overlapped with that of the northern Han Chinese.[110]  Estimated ancestry components among modern Eurasian populations. The red components represent the distinctive genetic markers characteristic of people with East Asian ancestry.[111] The genetic makeup of the modern Han Chinese is not purely uniform in terms of physical appearance and biological structure due to the vast geographical expanse of China and the migratory percolations that have occurred throughout it over the last three millennia. This has also engendered the emergence and evolution of the diverse multiplicity of assorted Han subgroups found throughout the various regions of modern China today. Comparisons between the Y chromosome single-nucleotide polymorphisms (SNPs) and mitochondrial DNA (mtDNA) of modern Northern Han Chinese and 3000 year old Hengbei ancient samples from China's Central Plains show that they are extremely similar to each other. These findings demonstrate that the core fundamental structural basis that shaped the genetic makeup of the present-day Northern Han Chinese was already formed three thousand years ago.[112] Studies of DNA remnants from the Central Plains area of China 3000 years ago show close affinity between that population and those of Northern Han today in both the Y-DNA and mtDNA. Both northern and southern Han show similar Y-DNA genetic structure.[113] Northern Han Chinese populations also have some West Eurasian admixture,[114] especially Han Chinese populations in Shaanxi (~2%-4.6%)[115] and Liaoning (~2%).[116] During the Zhou dynasty, or earlier, peoples with paternal haplogroup Q-M120 also contributed to the ethnogenesis of Han Chinese people. This haplogroup is implied to be widespread in the Eurasian steppe and north Asia since it is found among Cimmerians in Moldova and Bronze Age natives of Khövsgöl. But it is currently near-absent in these regions except for East Asia. In modern China, haplogroup Q-M120 can be found in the northern and eastern regions.[117] [118] Other Y-DNA haplogroups that have been found with notable frequency in samples of Han Chinese include O-P203 (15/165 = 9.1%, 217/2091 = 10.38%,[119] 47/361 = 13.0%), C-M217 (10/168 = 6.0%, 27/361 = 7.5%, 176/2091 = 8.42%,[119] 187/1730 = 10.8%, 20/166 = 12.0%), N-M231 (6/166 = 3.6%, 94/2091 = 4.50%,[119] 18/361 = 5.0%, 117/1729 = 6.8%, 17/165 = 10.3%), O-M268(xM95, M176) (78/2091 = 3.73%,[119] 54/1147 = 4.7%,[120] 8/168 = 4.8%, 23/361 = 6.4%, 12/166 = 7.2%), and Q-M242 (2/168 = 1.2%, 49/1729 = 2.8%, 61/2091 = 2.92%,[119] 12/361 = 3.3%, 48/1147 = 4.2%[120]). However, the mtDNA of Han Chinese increases in diversity as one looks from northern to southern China, which suggests that the influx of male Han Chinese migrants intermarried with the local female non-Han aborigines after arriving in what is now modern-day Guangdong, Fujian, and other regions of southern China.[121][122] Despite this, tests comparing the genetic profiles of northern Han, southern Han, and non-Han southern natives determined that haplogroups O1b-M110, O2a1-M88 and O3d-M7, which are prevalent in non-Han southern natives, were only observed in some southern Han Chinese (4% on average), but not in the northern Han genetic profile. Therefore, this proves that the male contribution of the southern non-Han natives in the southern Han genetic profile is limited, assuming that the frequency distribution of Y lineages in southern non-Han natives represents that prior to the expansion of Han culture which originated two thousand years ago from the north.[121][123] A recent, and to date the most extensive, genome-wide association study of the Han population, shows that geographic-genetic stratification from north to south has occurred and centrally placed populations act as the conduit for outlying ones.[124] Ultimately, with the exception in some ethnolinguistic branches of the Han Chinese, such as Pinghua and Tanka people,[125] there is a "coherent genetic structure" found in the entirety of the modern Han Chinese populace.[126] Although admixture proportions can vary according to geographic region, the average genetic distance between various Han Chinese populations is much lower than between European populations, for example.[127] Autosomal DNA A 2018 study calculated pairwise FST (a measure of genetic difference) based on genome-wide SNPs, among the Han Chinese (Northern Han from Beijing and Southern Han from Hunan, Jiangsu and Fujian provinces), Japanese and Korean populations sampled. It found that the smallest FST value was between Northern Han Chinese (Beijing) (CHB) and Southern Han (Hunan, Fujian, etc.) Chinese (CHS) (FST[CHB-CHS] = 0.0014), while CHB and Korean (KOR) (FST[CHB-KOR] = 0.0026) and between KOR and Japanese (JPT) (FST[JPT-KOR] = 0.0033). Generally, pairwise FST between Han Chinese, Japanese and Korean (0.0026~ 0.0090) are greater than that within Han Chinese (0.0014). These results suggested Han Chinese, Japanese and Korean are different in terms of genetic make-up, and the differences among the three groups are much larger than that between northern and southern Han Chinese.[128] Nonetheless, there is also genetic diversity among the Southern Han Chinese. The genetic composition of the Han population in Fujian might not accurately represent that of the Han population in Guangdong.  A PCA graph illustrates the genetic differences among Han Chinese groups.[129] Another study shows that the northern and southern Han Chinese are genetically close to each other and it finds that the genetic characteristics of present-day northern Han Chinese were already formed prior to three thousand years ago in the Central Plain area.[130] A recent genetic study on the remains of people (~4,000 years BP) from the Mogou site in the Gansu-Qinghai (or Ganqing) region of China revealed more information on the genetic contributions of these ancient Di-Qiang people to the ancestors of the Northern Han. It was deduced that 3,300 to 3,800 years ago some Mogou people had merged into the ancestral Han population, resulting in the Mogou people being similar to some northern Han in sharing up to ~33% paternal (O3a) and ~70% maternal (D, A, F, M10) haplogroups. The mixing ratio was possibly 13–18%.[131] The estimated contribution of northern Han to southern Han is substantial in both paternal and maternal lineages and a geographic cline exists for mtDNA. As a result, the northern Han are one of the primary contributors to the gene pool of the southern Han. However, it is noteworthy that the expansion process was not only dominated by males, as is shown by both contribution of the Y-chromosome and the mtDNA from northern Han to southern Han. Northern Han Chinese and Southern Han Chinese exhibit both Ancient Northern East Asian and Ancient Southern East Asian ancestries.[132] These genetic observations are in line with historical records of continuous and large migratory waves of northern China inhabitants escaping warfare and famine, to southern China. Aside from these large migratory waves, other smaller southward migrations occurred during almost all periods in the past two millennia.[133] A study by the Chinese Academy of Sciences into the gene frequency data of Han subpopulations and ethnic minorities in China showed that Han subpopulations in different regions are also genetically quite close to the local ethnic minorities, suggesting that in many cases, ethnic minorities ancestry had mixed with Han, while at the same time, the Han ancestry had also mixed with the local ethnic minorities.[134] Han Chinese, similar to other East Asian populations, have inherited West Eurasian ancestry, around 2.8% in Northern Han Chinese and around 1.7% in Southern Han Chinese.[135] An extensive, genome-wide association study of the Han population in 2008, shows that geographic-genetic stratification from north to south has occurred and centrally placed populations act as the conduit for outlying ones.[136] Ultimately, with the exception in some ethnolinguistic branches of the Han Chinese, such as Pinghua, there is "coherent genetic structure" (homogeneity) in all Han Chinese.[137] Paternal lineages The major haplogroups of Han Chinese belong to subclades of Haplogroup O-M175. Y-chromosome O2-M122 is a common DNA marker in Han Chinese, as it appeared in China in prehistoric times, and is found in more than 50% of Chinese males, with frequencies tending to be high toward the east of the country, ranging from 29.7% to 52% in Han from southern and central China, to 55–68% in Han from the eastern and northeastern Chinese mainland and Taiwan.[138] Other Y-DNA haplogroups that have been found with notable frequency in samples of Han Chinese include O-P203 (9.1–13.0%), C-M217 (6.0–12.0%), N-M231 (3.6–10.3%), O-M268(xM95, M176) (4.7–7%), and Q-M242 (2/168 = 1.2–4.2%).[120][138] Maternal lineages The mitochondrial-DNA haplogroups of the Han Chinese can be classified into the northern East Asian-dominating haplogroups, including A, C, D, G, M8, M9, and Z, and the southern East Asian-dominating haplogroups, including B, F, M7, N*, and R.[133] These haplogroups account for 52.7% and 33.85% of those in the Northern Han, respectively. Haplogroup D is the modal mtDNA haplogroup among northern East Asians. Among these haplogroups, D, B, F, and A were predominant in the Northern Han, with frequencies of 25.77%, 11.54%, 11.54%, and 8.08%, respectively. However, in the Southern Han, the northern and southern East Asian-dominating mtDNA haplogroups accounted for 35.62% and 51.91%, respectively. The frequencies of haplogroups D, B, F, and A reached 15.68%, 20.85%, 16.29%, and 5.63%, respectively.[130][139][140][141][142] Tibetan peoples Main article: Tibetan people The ethnic roots of Tibetans can be traced back to a deep Eastern Asian lineage representing the indigenous population of the Tibetan plateau since c. 40,000 to 30,000 years ago, and arriving Neolithic farmers from the Yellow River within the last 10,000 years associated, and which can be associated with having introduced the Sino-Tibetan languages. Modern Tibetans derive up to 20% from Paleolithic Tibetans, with the remaining 80% being primarily derived from Yellow River farmers. The present-day Tibetan gene pool was formed at least 5,100 years BP.[143][144] Paternal lineage Tibetan males predominantly belong to the paternal lineage D-M174 followed by lower amounts of O-M175.[145] Maternal lineage Tibetan females belong mainly to the Northeast Asian maternal haplogroups M9a1a, M9a1b, D4g2, D4i and G2ac, showing continuity with ancient middle and upper Yellow River populations.[146] Turkic peoples See also: Turkic peoples Linguistic and genetic evidence strongly suggests an early presence of Turkic peoples in eastern Mongolia.[147] The genetic evidence suggests that the Turkification of Central Asia was carried out by East Asian dominant minorities migrating out of Mongolia.[34] Genetic data found that almost all modern Turkic-speaking peoples retained at least some shared ancestry associated with "Southern Siberian and Mongolian" (SSM) populations, supporting this region as the "Inner Asian Homeland (IAH) of the pioneer carriers of Turkic languages" which subsequently expanded into Central Asia.[148] 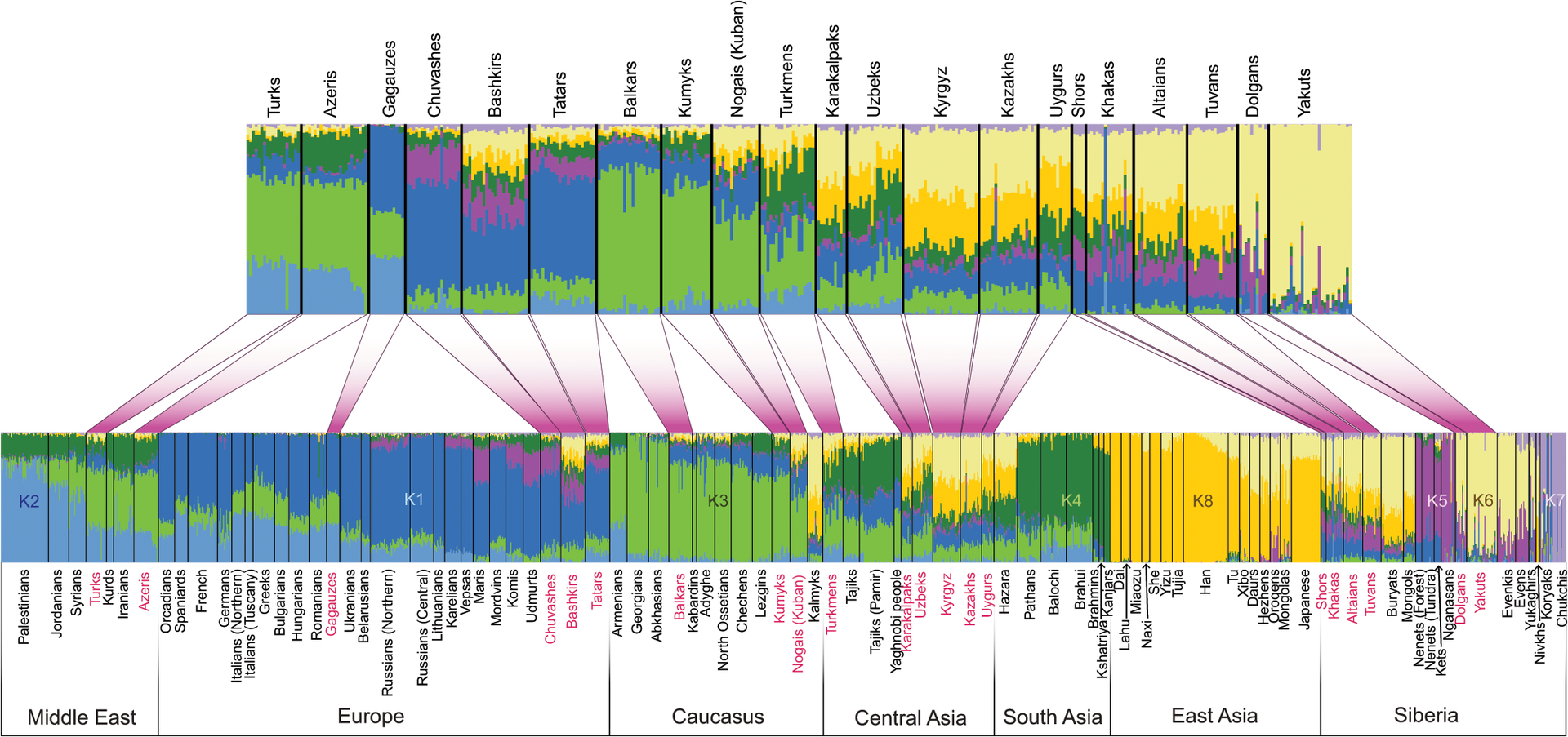 Population structure of Turkic-speaking populations in the context of their geographic neighbors across Eurasia. Turkic-speaking populations are shown in red. The upper barplot shows only Turkic-speaking populations. 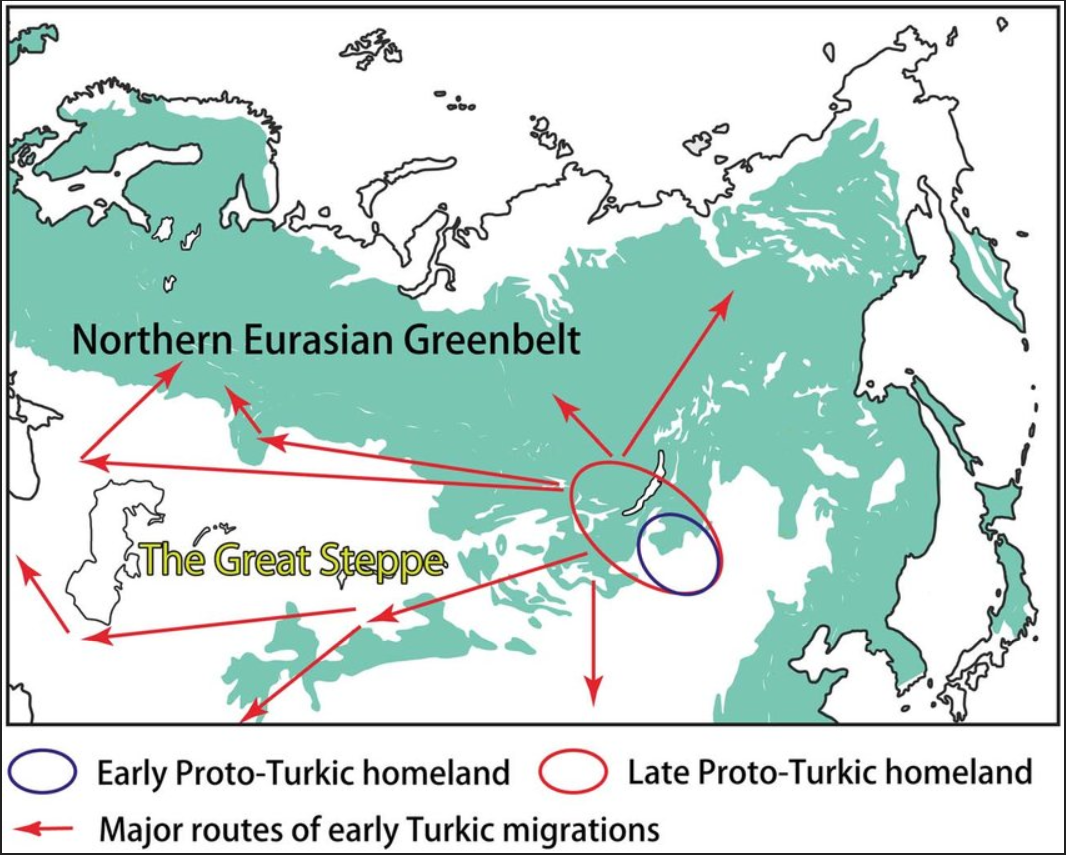 Genetic, archeologic and linguistic evidence links the early Turkic peoples with Northeast Asian millet-agriculturalists, which later adopted a nomadic lifestyle and expanded from eastern Mongolia westwards. An Ancient Northeast Asian origin of the early Turkic peoples has been corroborated in multiple recent studies. Early and medieval Turkic groups however exhibited a wide range of both (Northern) East Asian and West Eurasian genetic origins, in part through long-term contact with neighboring peoples such as Iranian, Mongolic, Tocharian, Uralic and Yeniseian peoples, and others.[149][150][151][152][153][154] Paternal lineages Common Y-DNA haplogroups in Turkic peoples are Haplogroup N-M231 (found with especially high frequency among Turkic peoples living in present-day Russia, especially among Siberian Tatars, as Zabolotnie Tatars have one of the highest frequencies of this haplogroup, second only to Samoyedic Nganasans ), Haplogroup C-M217 (especially in Central Asia, and in particular, Kazakhstan, also in Siberia among Siberian Tatars), Haplogroup Q-M242 (especially in Southern Siberia among the Siberian Tatars, also quite frequent among Lipka Tatars and among Turkmens and the Qangly tribe of Kazakhs), and Haplogroup O-M175 (especially among Turkic peoples living in present-day China, the Naiman tribe of Kazakhs and Siberian Tatars). Some groups also have Haplogroup R1b (notably frequent among the Teleuts, Siberian Tatars, and Kumandins of Southern Siberia, the Bashkirs of the Southern Ural region of Russia, and the Qypshaq tribe of Kazakhs), Haplogroup R1a (notably frequent among the Kyrgyz, Altaians, Siberian Tatars, Lipka Tatars, Volga Tatars, Crimean Tatars and several other Turkic peoples living in present-day Russia), Haplogroup J-M172 (especially frequent among Uyghurs, Azerbaijanis, and Turkish people), and Haplogroup D-M174 (especially among Yugurs, but also observed regularly with low frequency among Southern Altaians, Nogais, Kazakhs, and Uzbeks).[155][156] |
現代の人口 現代人と古代ユーラシア人の遺伝的構造[53] 詳細は「東アジアの人々」を参照 満州人とダウール人 詳細は「ツングース系民族」を参照 常染色体DNA 遼寧省の満州人に関する研究では、彼らは北部の漢民族と密接な遺伝的関係があり、混血の兆候が顕著であると報告されている。遼寧省の満州族は、黄河流域の 農耕民に由来する主要な祖先構成要素と、アムール川流域の古代民族に由来する、あるいはその他の古代民族に由来する小規模な祖先構成要素から形成されてい る。 満州族はツングース諸語を話す民族のまとまりのある遺伝的構造の例外であり、過去数百年間に大規模な人口移動と遺伝的混血があったためであると考えられ る。[54] 父系 多数のダウール人の男性がハプログループC-M217に属している(Xue Yali et al. 2006による12/39 = 30.8%[55]、Wang Chi-zao et 2018[56])、ハプログループO-M122は、現在のダウール族の間では2番目に多いハプログループである(10/39 = 25.6%、[55] 52/207 = 25.1%[56])。ダウール族の中には、主に他のY-DNAハプログループに属する部族(ハラ族、カザフ族の部族を参照)も存在し、ハプログループN -M46/M178(メルデン・ハラ族)やハプログループO1b1a1a-M95(ゴブル・ハラ族)などが挙げられる。[ 56] ハプログループC3b2b1*-M401(xF5483)[57][58][59] は、愛新覚羅家のマーカーとなりうるものとして特定されており、中国北部の10の異なる少数民族に存在しているが、漢民族にはほとんど存在していない。 [60][61][ 満州人もハプログループC-M217をかなりの割合で示すが、現代の満州人において最も多く見られるY-DNAハプログループはハプログループO- M122であり、これは中国の一般人口と共通している。 アイヌ民族 詳細は「アイヌ民族」を参照 初期のアイヌ民族の起源は不明であるが、一般的に縄文時代晩期のサツマ文化と関連があり、後にオホーツク文化の影響を受けたという見解が主流である。 [64] 遺伝子的には、アイヌ民族は縄文時代の日本人と最も近い関係にある。アイヌ民族の遺伝的構成は「東アジアの多様性の深い枝」を表している。現代の東アジア の人口と比較すると、アイヌ人は「北東シベリア人とより近い遺伝的関係」にある。[8][49] 日本人  集団遺伝学的な主成分分析(PCA)グラフ、東アジアの人口の亜構造を示す 詳細は「日本人」および「日本人に関する遺伝学および人体計測学的研究」を参照 現代の日本の日本人集団は、アイヌ人、琉球人、本土日本人(大和民族)という3つの別個の、しかし関連する人口統計にまで遡ることができる。これらの集団 は北東アジアで見られる集団と密接な関係があり[65][66][67]、アイヌ人は琉球人と最も類似しており[66][68]、大和人は他の東アジアの 人々の中で朝鮮人と最も類似している[69][70][71]。 常染色体DNA 日本人の遺伝的祖先の大半は、主に韓国人など他のアジア大陸のグループに関連する遺伝子に由来するが、残りの一部は、地元の縄文時代の狩猟採集民に由来する。  先史・原史時代の日本の文化変遷と並行する地理的変遷 全ゲノム解析によると、現代の日本人は北東アジア、東アジア、および縄文人の要素を保有している。 縄文人の狩猟採集民と弥生人の移住者に加え、弥生時代から古墳時代への変遷期に、韓国や中国との文化・政治的な親和性の強い新たな要素が導入されたという 仮説が立てられている。 父系 Y-DNAの多様性に関する世界的な包括的研究(Underhill et al. 2000)では、日本から23人の男性のサンプルが含まれており、そのうち35%がハプログループD-M174、26%がO-M175、22%がO- M122、 13%がC-M8とC-M130、4.3%がN-M128に属していた。[73] Poznikら(2016年)は、東京の日本人男性サンプルのハプログループを報告している。[74] 36%がD2-M17 9、32%がO2b-M176、18%がO3-M122、7.1%がC1a1-M8、3.6%がO2a-K18、3.6%がC2-M217であった。 母系系統 1000人ゲノム・プロジェクトの東京首都圏における日本人サンプルの分析によると、現代の日本人に見られるミトコンドリアDNAのハプログループは、D (35.6%)、B(13.6%)、M7(1 0.2%)、G(10.2%)、N9(8.5%)、F(7.6%)、A(6.8%)、Z(3.4%)、M9(2.5%)、M8(1.7%)である。 [76] 韓国人 詳細は「韓国人」を参照  地域参照パネル、主成分分析、混合解析 現代の韓国人は全体的に東南アジア人よりも北東アジア人に似ている。[77] 常染色体DNA 古代ゲノムの比較により、韓国人の遺伝的構成は、北東アジアの狩猟採集民と、長江流域から流入した稲作を行う東南アジアの農耕民との混血として最もよく説 明できることが明らかになった。[77] このことは、考古学的、歴史的、言語学的証拠によって裏付けられており、それらによると、 韓国人の直接の祖先は、新石器時代(紀元前8,000年~紀元前1,000年)および青銅器時代(紀元前1,500年~紀元前400年)に中国北東部およ び朝鮮半島に居住していた原韓国人であったということが、考古学的、歴史的、言語学的証拠から示唆されている。 かなりの遺伝的多様性の証拠がある[79]。初期の韓国南部の人々には縄文人の血統が高いレベルで存在している[80]。古代の韓国人が縄文人の血統を 失ったのは、中国北部からの移住者と混血を繰り返したためであるという仮説が立てられている[81]。三国時代には孤立した時代が続き、 現代の韓国人の遺伝子プールが均質化したという仮説である。[82][79] 2022年の研究では、現代の韓国人に縄文人としての遺伝子を有意に検出することはできなかったが、異なる祖先推定法を用いることで、現代の蔚山韓国人に は縄文人由来の遺伝子が3.1~4.4%含まれていることが分かった。しかし、この結果を生み出したモデルは最も信頼できるものではないと著者は示唆して いる。[83] 韓国人には、日本人と同様に、ミトコンドリアDNAおよびY染色体ハプログループの南方系および北方系の両方の証拠が観察されている。[53] 韓国人の現存する遺伝的多様性の70%以上は、鉄器時代のカンボジア人と血縁関係にある古代中国南部からの移民との混血によって説明できる。[84] 父系 ヒトのY染色体上の多型に関する研究では、これまでのところ、朝鮮民族が半島に次々と移住してきた人々とともに、主に同系婚を繰り返してきた独特な民族と して長い歴史を持っていることを示す証拠が得られている。また、Y染色体ハプログループも3つの主要グループに分類されている。[85] 韓国人の大半はハプログループO-M17 5(総数の約79%[78][86]で、うち約42%[86]から44%[78]がハプログループO2-M122、約31%[78]から32%[86]が ハプログループO1b2-M 176、および約2%[78]から3%[86]がハプログループO1a-M119に属している。一方、かなりの少数派がハプログループC2-M217の亜 系に属している(合計で約12%[78]から13%[86])。N-M231、D-M55、Q-M242などの他のY-DNAハプログループも、現代の韓 国人のごく一部に見られる。[87][88][89] 母系家系 韓国人のミトコンドリアDNA系統の研究では、ハプログループD4が最も高い頻度で、次いでハプログループB、そしてハプログループAとGの順であること が示されている。頻度が低いハプログループには、N9、Y、F、D5、M7、M8、M9、M10、M11、R11、C、Zがある。 モンゴル民族 詳細は「モンゴル民族」を参照 モンゴル民族の民族形成は、主に古代北東アジア人の拡大と関連している。その後、彼らは他の集団と接触するようになり、特に南方の漢民族や西方の西方草原 遊牧民と接触するようになった。モンゴル人の遊牧民としてのライフスタイルは、西の草原遊牧民から一部の影響を受けている可能性もあるが、両者の間には大 きな遺伝子の流れは見られず、文化の伝達を示唆している。[93][94] モンゴル人は、鮮卑と原始モンゴル人の子孫であると考えられている。前者はモンゴル人(ハルハ・モンゴル人とも呼ばれる)、オイラト、カルムイク人、南モ ンゴル人を含む。後者は、アバガ・モンゴル人、アバガナール人、アオハン人、バアリン人、ゴルロス・モンゴル人、ジャライド人、ジャルード人、キシギテン 人、フチド人、ムムヤン人、オニグド人からなる。ダウル人は、パラモンゴルのキタイ人の子孫である。[95] 父系 モンゴルおよびロシアのモンゴル人の大半はハプログループC-M217の亜系統に属し、[96] 次いでO-M175とN-M231が低い頻度で続いている。[97] 一部はハプログループQ-M242や、さまざまな西ユーラシアのハプログループに属している。[98] 母系系統 母系ハプログループは多様であるが、他の北アジアの集団と類似しており、ハプログループD、ハプログループC、ハプログループB、ハプログループAなど、 アメリカ先住民とアジアの集団に共通するものもある。西ユーラシアのミトコンドリアDNAハプログループは、少数派である。ハプログループHV、ハプログ ループU、ハプログループK、ハプログループI、ハプログループJはすべてモンゴロイドの人々にみられる。[100] 漢民族 関連項目:漢民族 漢民族は主に新石器時代の黄河農耕民の子孫であり、その祖先は主に古代北東アジア人で、古代東南アジア人の血も若干混ざっている。北方の漢民族は主に ANEAの祖先を持ち、ASEANの混血が中程度であるのに対し、南方の漢民族はANEAの祖先が依然として優勢であるものの、ASEANの祖先を北方の 漢民族よりもかなり多く持つ。 漢民族は、朝鮮人や大和人などの他の現代の東アジアの集団と密接な遺伝的関係がある。[103][104][105][106][107][108] [109] 2018年の研究論文では、漢民族は相関遺伝的関係において朝鮮人と大和人と密接に関連している一方で、遺伝的に容易に区別できることも分かった。そし て、同じ漢民族のサブグループは、遺伝的には韓国人と大和人よりも互いに近い関係にあるが、それでも互いに容易に区別できることが分かった。[109] 2020年に発表された研究では、大和人の人口は北部の漢民族と重複していることが分かった。[110]  現代のユーラシア系集団における推定される祖先構成。赤色の構成要素は、東アジア系の人々に特徴的な特有の遺伝子マーカーを表している。[111] 現代の漢民族の遺伝的構成は、中国が地理的に広大であること、および過去3千年間にわたって中国全土で起こった移住の浸透により、外見や生物学的構造の観 点では純粋に均一ではない。このことが、現代中国各地に存在するさまざまな漢民族のサブグループの多様性と進化を生み出した。現代の北方漢民族と、中国中 央平原から出土した3000年前の恒北古代サンプルのY染色体一塩基多型(SNP)とミトコンドリアDNA(mtDNA)を比較したところ、両者は極めて 類似していることが分かった。これらの調査結果は、現代の北方漢民族の遺伝的構成を形作った中核的かつ基本的な構造的基盤は、すでに3000年前に形成さ れていたことを示している。[112] 3000年前の中国中央平原地域から採取されたDNAの残存物の調査では、Y-DNAとmtDNAの両方において、その集団と現代の北方漢民族の集団との間に密接な類似性が認められた。北方漢民族と南方漢民族の両方で、Y-DNAの遺伝的構造は類似している。[113] 北方漢民族集団にも西ユーラシア混血が存在し[114]、特に陝西省(2%~4.6%)[115]と遼寧省(2%)[116]の漢民族集団にその傾向が強 い。 周王朝時代、あるいはそれ以前に、父系ハプログループQ-M120を持つ人々も漢民族の民族形成に貢献した。このハプログループは、モルドバのキンメリア 人や、ホブスゴル(Khövsgöl)の青銅器時代の原住民の間でも発見されているため、ユーラシア大陸の草原や北アジアに広く分布していたことが示唆さ れている。しかし、現在ではこれらの地域では東アジアを除いてほとんど見られない。現代の中国では、ハプログループQ-M120は北部および東部地域で見 られる。[117][118] 漢民族のサンプルで顕著な頻度で発見された他のY-DNAハプログループには、O-P203(15/165 = 9.1%、217/2091 = 10.38%、[1 19] 47/361 = 13.0%)、C-M217 (10/168 = 6.0%, 27/361 = 7.5%, 176/2091 = 8.42%, [119] 187/1730 = 10.8%, 20/ 166 = 12.0%), N-M231 (6/166 = 3.6%, 94/2091 = 4.50%, [119] 18/361 = 5.0%, 117/1729 = 6.8%, 17/165 = 10.3%), O-M268(xM95, M176) (78/2091 = 3.73%, [119] 54/1147 = 4.7%, [120] 8/168 = 4.8%, 23/361 = 6.4%, 12/166 = 7.2%)、Q-M242(2/168 = 1.2%、49/1729 = 2.8%、61/2091 = 2.92%、[119] 12/361 = 3.3%、48/1147 = 4.2%[120])。 しかし、中国人のミトコンドリアDNAは、中国北部から南部に向かうにつれて多様性が増している。これは、現代の広東省、福建省、および中国南部の他の地 域に到着した男性の漢民族移民が、現地の非漢民族の女性と結婚したことを示唆している。[121][122] にもかかわらず、 北方漢族、南方漢族、および漢族以外の南方土着民の遺伝子プロファイルを比較したところ、漢族以外の南方土着民に多いハプログループO1b-M110、 O2a1-M88、O3d-M7は、南方漢族の一部(平均4%)にのみ見られ、北方漢族の遺伝子プロファイルには見られないことが判明した。したがって、 南部の非漢民族の男性が南部の漢民族の遺伝的プロフィールに与える影響は限定的であることが証明される。南部の非漢民族におけるY遺伝子系列の頻度分布 が、2000年前に北方から始まった漢文化の拡大以前の状況を表していると仮定すると、このことが証明される。[121][123] 最近の、そして現在までで最も広範囲にわたる漢民族のゲノムワイド関連解析では、北部から南部にかけて地理的・遺伝的階層化が起こっており、中心部に位置 する集団が周辺部の集団の伝導体として機能していることが示されている。[124] 最終的には、ピン 華語やタンカ族など、漢民族の一部の民族語派を除いては、[125] 現代の漢民族全体に「一貫した遺伝的構造」が認められる。[126] 混血の割合は地理的地域によって異なるが、さまざまな漢民族集団間の平均遺伝的距離は、例えばヨーロッパの集団間の距離よりもはるかに低い。[127] 常染色体DNA 2018年の研究では、サンプル採取した漢民族(北京出身の北方漢民族と、湖南省、江蘇省、福建省出身の南方漢民族)、日本人、韓国人の集団を対象に、ゲ ノム全体のSNPを基に、ペアワイズFST(遺伝的差異の指標)を算出した。最も小さいFST値は、北方漢民族(北京)(CHB)と南方漢民族(湖南、福 建など)(CHS)との間(FST[CHB-CHS]=0.0014)で、 一方、CHBと韓国人(KOR)(FST[CHB-KOR]=0.0026)の間、およびKORと日本人(JPT)(FST[JPT-KOR]= 0.0033)の間では、それぞれ0.0026、0.0033であった。一般的に、漢民族、日本人、韓国人の間の対立遺伝子間距離(FST) (0.0026~0.0090)は、漢民族内の対立遺伝子間距離(0.0014)よりも大きい。これらの結果は、漢民族、日本人、韓国人は遺伝子的に異な ることを示唆しており、3つのグループ間の違いは、漢民族の北部と南部間の違いよりもはるかに大きいことを示している。[128] とはいえ、南部の漢民族の間にも遺伝的多様性がある。福建省の漢民族の遺伝的構成は、広東省の漢民族の遺伝的構成を正確に反映していない可能性がある。  主成分分析(PCA)のグラフは、漢民族グループ間の遺伝的相違を示している。[129] 別の研究では、北方と南方の漢民族は遺伝的に近いことが示されており、現在の北方漢民族の遺伝的特性は、3000年以上前の中国中央平原地域においてすでに形成されていたことが分かっている。[130] 中国甘粛省と青海省の境界にあるモウコウ遺跡から出土した人骨(約4,000年前)の遺伝子を調査した最近の研究では、古代のディ・チアン族が北方漢民族 の祖先に対して遺伝的に与えた影響について、さらに詳しい情報が明らかになった。3,300年から3,800年前に、一部のモゴウ人が漢民族の祖先と融合 した結果、モゴウ人は父系で最大33%(O3a)、母系で最大70%(D、A、F、M10)のハプログループを共有する一部の北方漢人と類似するように なったと推定される。混合比率は13~18%であった可能性がある。[131] 北方漢人の南方漢人への貢献は父系および母系の系統の両方で相当なものであり、ミトコンドリアDNAには地理的な勾配が存在する。その結果、北方漢人は南 方漢人の遺伝子プールに大きく貢献している。しかし、北部漢族から南部漢族へのY染色体とミトコンドリアDNAの寄与率の両方から示されるように、その拡 大プロセスは男性だけによるものではなかったことは注目に値する。北部漢族と南部漢族は、古代の北東アジアと古代の南東アジアの祖先を持つ。[132] これらの遺伝的観察結果は、戦乱や飢饉を逃れて中国北部の住民が中国南部へ大規模な移住を繰り返したという歴史的記録と一致している。こうした大規模な移 住の波とは別に、過去2千年間のほぼすべての時代において、より小規模な南への移住も発生していた。[133] 中国科学院による、中国国内の漢民族の亜集団および少数民族の遺伝子頻度データに関する研究では、 各地域の漢民族集団は、遺伝子的にその地域の少数民族と非常に近いことも示しており、多くの場合、少数民族の祖先が漢民族と混血したと同時に、漢民族の祖 先もその地域の少数民族と混血したことを示唆している。[134] 漢民族は他の東アジアの民族と同様に、西ユーラシアの祖先を継承しており、北方の漢民族では約2.8%、南方の漢民族では約1.7%である。[135] 2008年の漢民族を対象とした広範囲にわたるゲノムワイド関連解析では、地理的・遺伝的層別化が北部から南部にかけて生じていることが示され、中心部に 位置する集団が周辺部の集団の媒介となっていることが明らかになった。[136] 最終的には、ピンフア語のような漢民族のいくつかの民族言語の分派を除いて、すべての漢民族に「一貫した遺伝的構造」(均質性)がある。[137] 父系 漢民族の主要なハプログループはハプログループO-M175の亜系に属する。Y染色体O2-M122は、先史時代に中国で出現したもので、漢民族の一般的 なDNAマーカーであり、中国人の男性の50%以上に見られ、その頻度は 中国東部ではその頻度は高く、華南および華中では29.7%から52%の漢民族に、中国東部および中国本土北東部および台湾では55%から68%の漢民族 に見られる。[138] 漢民族のサンプルで顕著な頻度で発見された他のY-DNAハプログループには、O-P203(9.1~13.0%)、C-M217 (6.0~12.0%)、N-M231(3.6 10.3%)、O-M268(xM95, M176) (4.7–7%)、Q-M242 (2/168 = 1.2–4.2%) などである。[120][138] 母系 漢民族のミトコンドリアDNAハプログループは、A、C、D、G、M8、M9、Zなどの北東アジア優勢ハプログループと、B、F、M7、N*、Rなどの南東アジア優勢ハプログループに分類される。 これらのハプログループは、それぞれ北方漢民族の52.7%と33.85%を占めている。ハプログループDは、北方東アジア人における最も一般的なミトコ ンドリアDNAハプログループである。これらのハプログループのうち、D、B、F、Aは北部漢民族に多く、それぞれ25.77%、11.54%、 11.54%、8.08%の頻度であった。 しかし、南部漢民族では、東アジア優勢のハプログループのうち、北部と南部のハプログループがそれぞれ35.62%と51.91%を占めた。ハプログルー プD、B、F、Aの頻度はそれぞれ15.68%、20.85%、16.29%、5.63%に達した。[130][139][140][141][142] チベット民族 詳細は「チベット民族」を参照 チベット人の民族的な起源は、約4万年前から3万年前のチベット高原の先住民に遡る東アジアの系統にまで遡ることができ、さらに1万年前には黄河から新石 器時代の農耕民が到来し、それらが結びついた。現代のチベット人の遺伝子は、旧石器時代のチベット人から最大20%、残りの80%は主に黄河農耕民から派 生している。現代のチベット人の遺伝子プールは、少なくとも5,100年前に形成された。[143][144] 父系 チベット人男性は、父系D-M174に属する者が大半を占め、次いでO-M175が少ない。[145] 母系 チベット人女性は、主に北東アジアの母系ハプログループM9a1a、M9a1b、D4g2、D4i、G2acに属し、古代の中部および上流の黄河流域の集団との連続性を示している。[146] トルコ系民族 関連項目:トルコ系民族 言語学的および遺伝学的証拠は、モンゴル東部にトルコ系民族が早くから存在していたことを強く示唆している。[147] 遺伝学的証拠は、中央アジアのトルコ化はモンゴルから移住した東アジアの支配的少数派によって行われたことを示唆している。[34] 遺伝子データによると、現代のトルコ語話者のほぼすべてが、「南シベリア・モンゴル系(SSM)」の集団と少なくとも何らかの共通の祖先を持つことが判明 しており、この地域が「トルコ語を話す人々の先駆的担い手による内アジアの故郷(IAH)」であり、その後中央アジアに拡大したことを裏付けている。  ユーラシア大陸の地理的近隣地域におけるトルコ語話者集団の人口構造。トルコ語話者集団は赤で示されている。上部の棒グラフはテュルク語話者集団のみを示す。  遺伝学的、考古学的、言語学的証拠から、初期のトルコ系民族は北東アジアのキビ農耕民と関連があり、後に遊牧生活を営むようになり、モンゴル東部から西へと拡大していった。 初期のトルコ系民族の古代北東アジア起源は、複数の最近の研究で裏付けられている。しかし、初期および中世のトルコ系民族は、(北)東アジアと西ユーラシ アの双方の遺伝的起源を幅広く示しており、その理由の一部は、イラン系民族、モンゴル系民族、トカラ系民族、ウラル系民族、エニセイ系民族など、近隣民族 との長期にわたる接触によるものである。[149][150][151][152][153][154] 父系家系 トルコ系民族における一般的なY-DNAハプログループは、ハプログループN-M231(特に現在のロシアに住むトルコ系民族の間で高い頻度で発見されて おり、特にシベリア・タタール人の間で高い頻度で発見されている。ザボロトニー・タタール人はこのハプログループの頻度が最も高く、サモエド人のンガナサ ン人に次いで2番目に高い)、ハプログループC-M217(特に中央アジア、特にカザフスタンで 、またシベリア・タタール人の間でもシベリア南部で特に多い)、ハプログループQ-M242(特にシベリア・タタール人の間で南シベリアに多い。また、リ プカ・タタール人やトルクメン人、カザフ人のカングル部族の間でもかなり多い)、ハプログループO-M175(特に現在の中国に住むトルコ系民族、カザフ 人のナイマン部族、シベリア・タタール人の間で多い)である。また、ハプログループR1b(テレウト族、シベリア・タタール族、南シベリアのクマンディン 族、ロシア南ウラル地方のバシキール族、カザフ族のキプシャク族に特に多く見られる)、ハプログループR1a(キルギス族、アルタイ族、シベリア・タター ル族、リプカ・タタール族、ボルガ・タタール族、 クリミア・タタール人、および現在のロシアに住む他のいくつかのトルコ系民族)、ハプログループJ-M172(特にウイグル人、アゼルバイジャン人、トル コ人に多い)、ハプログループD-M174(特にユグール人に多いが、南アルタイ人、ノガイ人、カザフ人、ウズベク人にも低頻度で定期的に観察される) [155][156] |
| Relationship to other Asia-Pacific and Native American populations Central Asians See also: History of Central Asia § Medieval 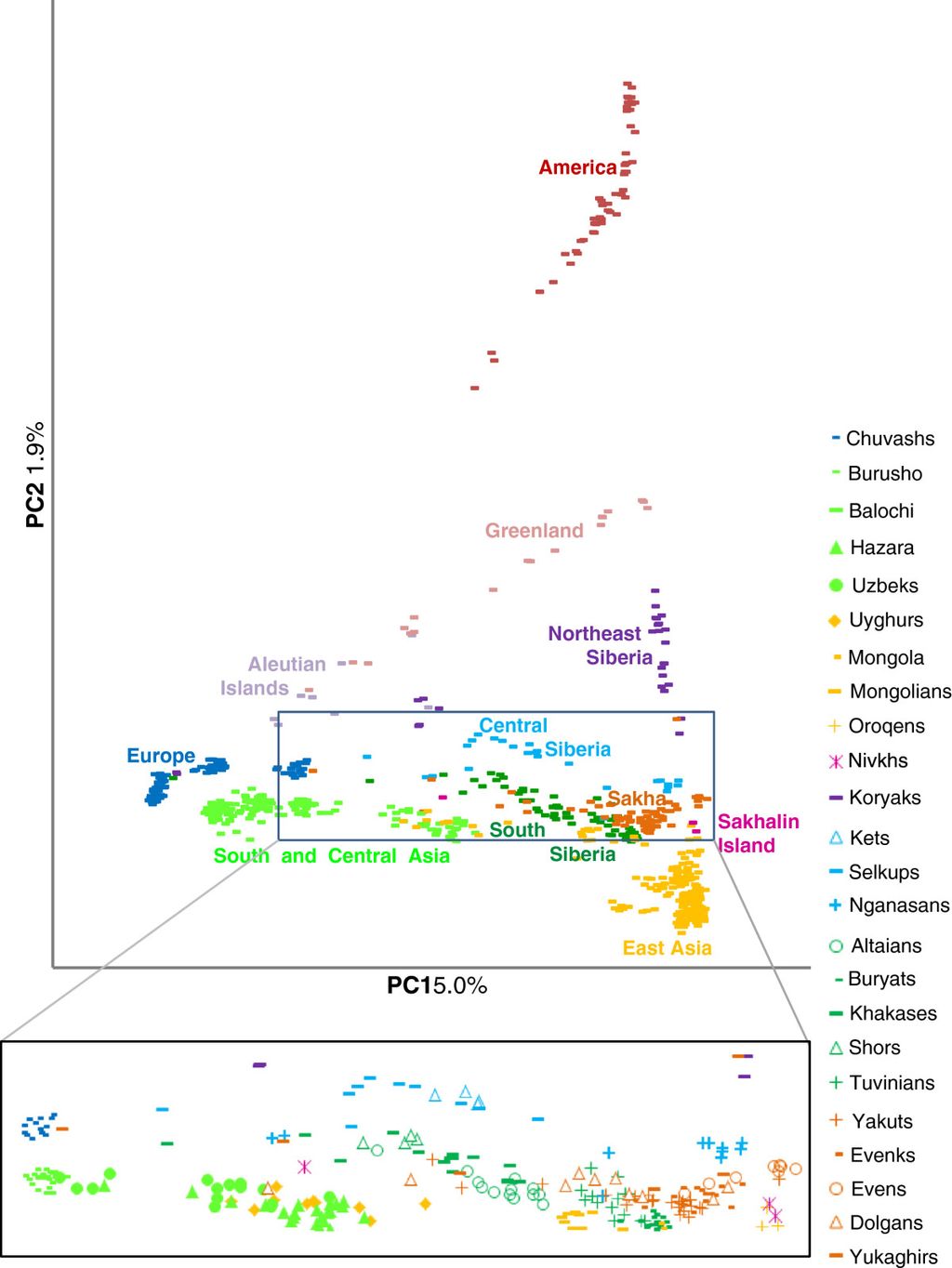 PCA of various populations in the context of Eurasia and the Americas The genetic evidence suggests that the Turkification of Central Asia was carried out by East Asian dominant minorities migrating out of Mongolia.[157] According to a recent study, the Turkic Central Asian populations, such as Kyrgyz, Kazakhs, Uzbeks, and Turkmens share more of their gene pool with various East Asian and Siberian populations than with West Asian or European populations. The study further suggests that both migration and linguistic assimilation helped to spread the Turkic languages in Eurasia.[158] North Asians and Native Americans Genetic data suggests that Siberia was populated during the Terminal Upper-Paleolithic (36±1.5ka) period from a distinct Paleolithic population migrating through Central Asia into Northern Siberia. This population is known as Ancient North Eurasians or Ancient North Siberians. Between 30,000 and 25,000 years ago, the ancestors of both Paleo-Siberians and Native Americans originated from admixture between Ancient North Eurasians/Siberians and an Ancient East Asian lineage.[159][160] Ancestral Native Americans (or Ancient Beringians) later migrated towards the Beringian region, became isolated from other populations, and subsequently populated the Americas. Further geneflow from Northeast Asia resulted in the modern distribution of "Neo-Siberians" (associated with 'Altaic speakers') through the merger of Paleo-Siberians with Northeast Asians.[161][162][163] Overall, while Northern Asians cluster closely to East Asians, they are shifted into a distinct position. "Analyses of all 122 populations confirm many known relationships and show that most populations from North Asia form a cluster distinct from all other groups. Refinement of analyses on smaller subsets of populations reinforces the distinctiveness of North Asia and shows that the North Asia cluster identifies a region that is ancestral to Native Americans."[164] Native Americans Multiple studies suggests that all Native Americans ultimately descended from a single founding population that initially diverged from" Ancestral Beringians" which shared a common origin with Paleo-Siberians from the merger of Ancient North Eurasians and a Basal-East Asian source population in Mainland Southeast Asia around 36,000 years ago, at the same time at which the proper Jōmon people split from Basal-East Asians, either together or during a separate expansion wave. The basal northern and southern Native American branches, to which all other Indigenous peoples belong, diverged around 16,000 years ago, although earlier dates were also proposed.[53][165] An indigenous American sample from 16,000 BCE in Idaho, which is craniometrically similar to modern Native Americans, was found to have been closely related to Paleosiberians, confirming that Ancestral Native Americans split from an ancient Siberian source population somewhere in northeastern Siberia. Genetic data on samples with alleged "Paleo-Indian" morphology turned out to be closely related to contemporary Native Americans, disproving a hypothetical earlier migration into the Americas. The scientists suggest that variation within Native American morphology is just that, the natural variation which have arisen during the formation of Ancestral Native Americans. Signals of a hypothetical "population Y", if not a false positive, are likely explained through a now extinct population from East Asia (e.g. Tianyuan man, which contributed low amounts of ancestry to the Ancestral Native American gene pool in Asia, and perhaps also towards other Asian and Oceanian populations.[166][165][167][168][169] South Asians Main article: Genetic history of South Asia The genetic makeup of modern South Asians can be described as a combination of West Eurasian ancestries with divergent East Eurasian ancestries. The latter primarily include an indigenous South Asian component (termed Ancient Ancestral South Indians, short "AASI") that is distantly related to the Andamanese peoples, as well as to East Asians and Aboriginal Australians, and further include additional, regionally variable East/Southeast Asian components.[170][5] The East Asian-related ancestry component forms the major ancestry among Tibeto-Burmese and Khasi-Aslian speakers in the Himalayan foothills and Northeast India,[171][172] and is generally distributed throughout South Asia at lower frequency, with substantial presence in Mundari-speaking groups.[171][172] According to a genetic research (2015) including linguistic analyses, suggests an East Asian origin for proto-Austroasiatic groups, which first migrated to Southeast Asia and later into India.[173] According to Ness, there are three broad theories on the origins of the Austroasiatic speakers, namely northeastern India, central or southern China, or southeast Asia.[174] Multiple researches indicate that the Austroasiatic populations in India are derived from (mostly male dominated) migrations from Southeast Asia during the Holocene.[175][173][176][177][178][179] According to Van Driem (2007), "...the mitochondrial picture indicates that the Munda maternal lineage derives from the earliest human settlers on the Subcontinent, whilst the predominant Y chromosome haplogroup argues for a Southeast Asian paternal homeland for Austroasiatic language communities in India."[176]: 7 According to Chaubey et al. (2011), "Austroasiatic speakers in India today are derived from dispersal from Southeast Asia, followed by extensive sex-specific admixture with local Indian populations."[175] According to Zhang et al. (2015), Austroasiatic (male) migrations from southeast Asia into India took place after the lates Glacial maximum, circa 4,000 years ago.[173] According to Arunkumar et al. (2015), Y-chromosomal haplogroup O2a1-M95, which is typical for Austroasiatic speaking peoples, clearly decreases from Laos to east India, with "a serial decrease in expansion time from east to west," namely "5.7 ± 0.3 Kya in Laos, 5.2 ± 0.6 in Northeast India, and 4.3 ± 0.2 in East India." This suggests "a late Neolithic east to west spread of the lineage O2a1-M95 from Laos."[178][180] According to Riccio et al. (2011), the Munda people are likely descended from Austroasiatic migrants from southeast Asia.[177][181] According to Ness, the Khasi probably migrated into India in the first millennium BCE.[174] According to Yelmen et al. 2019, the two main components of Indian genetic variation; the South Asian populations that "separated from East Asian and Andamanese populations" form one of the deepest splits among non-African groups compared to the West Eurasian component because of "40,000 years of independent evolution".[182] Geneflow from Southeast Asians (particularly Austroasiatic groups) to South Asian peoples is associated with the introduction of rice-agriculture to South Asia. There is significant cultural, linguistic, and political Austroasiatic influence on early India, which can also be observed by the presence of Austroasiatic loanwords within Indo-Aryan languages.[183][184] Southeast Asians  Estimated ancestry components among selected modern populations per Changmai et al. (2022). The yellow component represents East Asian-like ancestry.[185] A 2020 genetic study about Southeast Asian populations, found that mostly all Southeast Asians are closely related to East Asians and have mostly "East Asian-related" ancestry.[20][186] Ancient remains of hunter-gatherers in Maritime Southeast Asia, such as one Holocene hunter-gatherer from South Sulawesi, had ancestry from both, an Australasian lineage (represented by Papuans and Aboriginal Australasians) and an "Ancient Asian" lineage (represented by East Asians or Andamanese Onge). The hunter-gatherer individual had approximately c. 50% "Basal-East Asian" ancestry and c. 50% Australasian/Papuan ancestry, and was positioned in between modern East Asians and Papuans of Oceania. The authors concluded that East Asian-related ancestry expanded from Mainland Southeast Asia into Maritime Southeast Asia much earlier than previously suggested, as early as 25,000 BCE, long before the expansion of Austroasiatic and Austronesian groups.[187] A 2022 genetic study confirmed the close link between East Asians and Southeast Asians, which the authors term "East/Southeast Asian" (ESEA) populations, and also found a low but consistent proportion of South Asian-associated "SAS ancestry" (best samplified by modern Bengalis from Dhaka, Bangladesh) among specific Mainland Southeast Asian (MESA) ethnic groups (~2–16% as inferred by qpAdm), likely as a result of cultural diffision; mainly of South Asian merchants spreading Hinduism and Buddhism among the Indianized kingdoms of Southeast Asia. The authors however caution that Bengali samples harbor detechtable East Asian ancestry, which may affect the estimation of shared haplotypes. Overall, the geneflow event is estimated to have happened between 500 and 1000 YBP.[188] 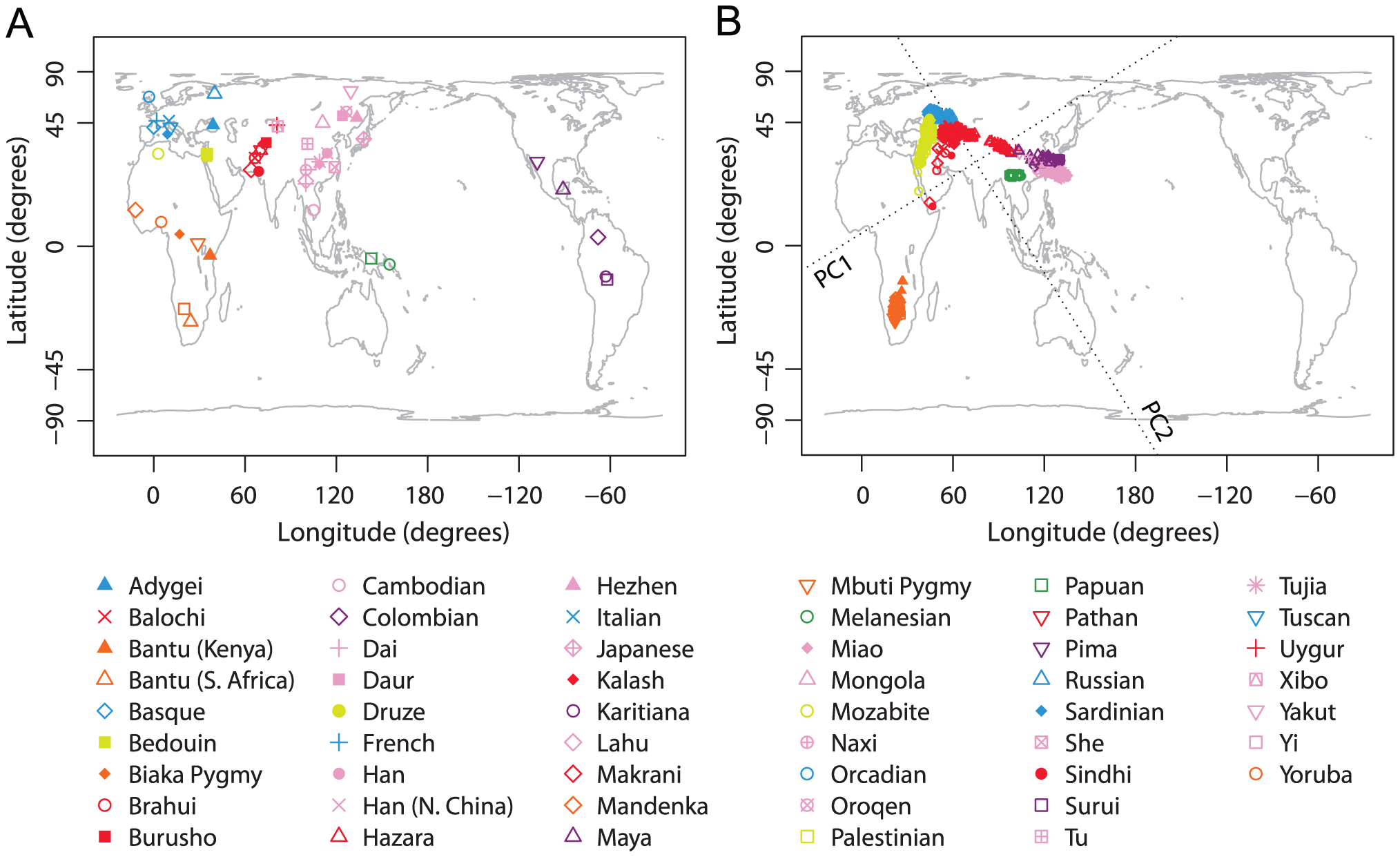 PCA plot of genetic variationof worldwide populations. Australasians (green) cluster relative close to other East Eurasians, such as East/Southeast Asians. Australasians Main articles: Indigenous people of New Guinea, Melanesians, and Aboriginal Australians Melanesians and Aboriginal Australians are deeply related to East Asians. Genetic studies have revealed that Australasians descended from the same Eastern Eurasian source population as East Asians and indigenous South Asians (AASI).[5] |
他のアジア太平洋およびネイティブアメリカンの集団との関係 中央アジア人 参照:中央アジアの歴史 § 中世  ユーラシアとアメリカ大陸における様々な民族集団の主成分分析 遺伝学的証拠によると、中央アジアのトルコ化はモンゴルから移住してきた東アジアの支配的少数派によって行われたことが示唆されている。[157] 最近の研究によると、キルギス人、カザフ人、ウズベク人、トルクメン人などの中央アジアのトルコ系民族は、西アジアやヨーロッパの民族よりも、むしろ様々 な東アジアやシベリアの民族と遺伝子プールを共有している。この研究はさらに、移動と言語的同化の両方が、ユーラシアにおけるテュルク諸語の拡散に役立っ たことを示唆している。[158] 北アジア人とネイティブアメリカン 遺伝子データによると、シベリアには、中央アジアを経由して北シベリアに移住した独特の旧石器時代の人々によって、後期旧石器時代(36±1.5ka)に人口が増加した。この集団は、古代北ユーラシア人または古代北シベリア人として知られている。 3万~2万5千年前、古代北ユーラシア人/シベリア人と古代東アジア系の人々の混血から、旧シベリア人とアメリカ先住民の祖先が生まれた。[159] [160] アメリカ先住民の祖先(または古代ベーリング人)はその後ベーリング地域へ移住し、他の集団から孤立し、その後アメリカ大陸に定住した。北東アジアからの さらなる遺伝子の流れは、旧シベリア人と北東アジア人の融合により、「ネオ・シベリア人」(「アルタイ語話者」に関連する)の現代的な分布をもたらした。 全体として、北方アジア人は東アジア人に近い位置に集まっているが、明確に異なる位置にシフトしている。「122の集団すべてについての分析により、多く の既知の関係が確認され、北アジアのほとんどの集団が他のすべての集団とは異なるクラスターを形成していることが示された。集団のより小さなサブセットに ついての分析の精緻化により、北アジアの独自性がさらに強調され、北アジアのクラスターがネイティブアメリカンの祖先である地域を特定していることが示さ れた」[164] ネイティブアメリカン 複数の研究が、ネイティブアメリカンはすべて、最終的には単一の祖先集団から分岐したと示唆している。その祖先集団は、約3万6千年前に古代ユーラシア人 と東アジアの基底集団が融合して誕生した旧シベリア人と共通の起源を持つ「先祖ベーリング人」から、最初に分岐した 約3万6千年前に東南アジア本土で古代北ユーラシア人と基底東アジア人の集団が融合したことによる。この時期は、縄文人たちが基底東アジア人から分岐した 時期と同じであり、同時に、あるいは別の拡散波の中で分岐した可能性もある。他のすべての先住民が属する、基底的な北米および南米のネイティブアメリカン 分岐は、約1万6000年前に分岐した。ただし、それより早い年代も提案されている。[53][165] 1万6000年前のアイダホ州から出土したアメリカ先住民のサンプルは、 現代のネイティブアメリカンと頭蓋計測学的に類似しているアイダホ州の紀元前16,000年のサンプルは、パレオシベリア人と密接な関係にあることが判明 し、先祖のネイティブアメリカンがシベリア北東部のどこかで古代のシベリア人集団から分岐したことが確認された。旧石器時代人」の形態を持つとされるサン プルの遺伝子データは、現代のネイティブアメリカンと密接な関係にあることが判明し、アメリカ大陸への仮説上の早期移住説は否定された。科学者たちは、ネ イティブアメリカンの形態の多様性は、単に祖先的ネイティブアメリカンの形成中に生じた自然な多様性であると示唆している。仮説上の「集団Y」の兆候は、 偽陽性でないとすれば、現在では絶滅している東アジアの集団(例えば、アジアにおける祖先的ネイティブアメリカンの遺伝子プールにわずかな量の祖先性を与 えた天元人、また、おそらくは他のアジアおよびオセアニアの集団にも)によって説明できる可能性が高い。[166][165][167][168] [169] 南アジア人 詳細は「南アジアの遺伝的歴史」を参照 現代の南アジア人の遺伝的構成は、西ユーラシアの祖先と東ユーラシアの祖先の組み合わせと表現することができる。後者は主に、アンダマン諸島の人々や東ア ジア人、オーストラリアのアボリジニと遠縁の関係にある南アジアの先住民(古代南インド人、略称「AASI」)の要素を含み、さらに地域によって異なる東 アジア人/東南アジア人の要素も含む。 関連祖先成分は、ヒマラヤ山麓およびインド北東部におけるチベット・ビルマ語族およびカシ・アスリア語族話者の間で主要な祖先成分を形成しており [171][172]、南アジア全体に低頻度で広く分布しており、ムンダリ語派話者グループにかなりの存在感を示している[171][172]。 言語分析を含む遺伝学の研究(2015年)によると、オーストロアジア語族の祖語は東アジア起源であり、最初に東南アジアへ、後にインドへと移住したこと が示唆されている。[173] ネスによると、オーストロアジア語族の起源については、大きく分けて3つの説があり、すなわちインド北東部 インド北東部、中国中部または南部、あるいは東南アジアである。複数の研究が、インドのオーストロアジア語族系民族は、完新世に東南アジアから(主に男性 が中心となった)移住してきた人々から派生したものであることを示している。ヴァン・ドリーム(2007年)によると、「ミトコンドリアの分析結果では、 ムンダー語族の母系は亜大陸に最初に移住した人々から派生したことが示されている。一方、優勢なY染色体ハプログループは、インドにおけるオーストロアジ ア語族の言語コミュニティの父系は東南アジアに起源を持つことを示唆している」[176]:7 Chaubey ら(2011年)によると、「現在のインドにおけるオーストロアジア語族話者は、東南アジアからの分散に由来し、その後、インドの地元住民との広範囲にわ たる性別ごとの混血が続いた」[175]。Zhang ら(2015年)によると、 東南アジアからインドへのオーストロアジア語族(男性)の移住は、最終氷期最盛期以降、約4000年前に起こった。[173] Arunkumar et al. (2015) によると、 オーストロアジア語族を話す人々に典型的なY染色体ハプログループO2a1-M95は、ラオスから東インドにかけて明らかに減少しており、「東から西への 拡散時間の連続的な減少」、すなわち「ラオスでは5.7±0.3 Kya、北東インドでは5.2±0.6、東インドでは4.3±0.2」が認められる。これは「ラオスから東西に広がった新石器時代後期のO2a1-M95 系統」を示唆している。[178][180] リッチオら(2011年)によると、ムンダー人は東南アジアからのオーストロアジア語族の移住者から派生した可能性が高い。[177][181] ネスによると、カシ人はおそらく紀元前1千年紀にインドに移住した。[174] Yelmenら(2019年)によると、インドの遺伝的多様性の2つの主な要素である「東アジアおよびアンダマン諸島の集団から分岐した」南アジアの集団 は、「4万年にわたる独自の進化」により、西ユーラシアの要素と比較して、非アフリカ集団の中で最も深い分岐の1つを形成している。 東南アジア人(特にオーストロアジア語族)から南アジアの人々への遺伝子の流れは、南アジアへの稲作農業の伝来と関連している。初期のインドには、文化、 言語、政治の面でオーストロアジア語族の影響が顕著に見られ、それはインド・アーリア語族の言語の中にオーストロアジア語族からの借用語が存在することか らも観察できる。[183][184] 東南アジア人  Changmai et al. (2022)による、選択された現代集団における推定される祖先構成要素。黄色の要素は東アジア系の祖先を表す。[185] 2020年の東南アジアの集団に関する遺伝学的研究では、ほとんどの東南アジア人は東アジア人と密接に関連しており、ほとんどが「東アジア系」の祖先を持つことが分かった。[20][186] 南スラウェシの完新世の狩猟採集民など、東南アジアの海洋地域における古代の狩猟採集民の遺跡は、オーストラレーシア系(パプア人やオーストラリアのアボ リジニに代表される)と「古代アジア人」系(東アジア人やアンダマン諸島のオンゲ族に代表される)の両方の祖先を持っていた。この狩猟採集民は、約50% が「基底東アジア人」の血筋で、約50%がオーストラレーシア人/パプア人の血筋であり、現代の東アジア人とオセアニアのパプア人の間に位置づけられる。 著者らは、東アジア関連の祖先が東南アジア大陸部から東南アジアの島嶼部へと拡大したのは、これまでの推定よりもはるかに早く、紀元前25,000年頃、 オーストロアジア語族やオーストロネシア語族の拡散よりもずっと前のことだったと結論づけた。[187] 2022年の遺伝学的研究では、東アジア人と東南アジア人の密接なつながりが確認され、著者はこれを「東・東南アジア人(ESEA)」と名付けた。また、 特定の東南アジア大陸部(MESA)の民族集団の間では、南アジアに関連する「SAS遺伝子」の割合が低いが一定していることも分かった( 主に南アジアの商人たちが、インド化された東南アジアの王国の間でヒンドゥー教や仏教を広めた結果であると考えられる。ただし、ベンガル人のサンプルには 分離可能な東アジアの祖先が含まれている可能性があり、共有ハプロタイプの推定に影響を与える可能性があるという注意が著者の側からなされている。全体と して、遺伝子交流の出来事は500年から1000年前のものと推定されている。[188]  世界中の集団の遺伝的多様性の主成分分析プロット。オーストラリア人(緑)は、東アジア人や東南アジア人などの他の東ユーラシア人と比較的近い位置に集まっている。 オーストラリア人 詳細は:ニューギニアの先住民、メラネシア人、オーストラリアのアボリジニ メラネシア人とオーストラリアのアボリジニは東アジア人と深い関係にある。遺伝学的研究により、オーストラリア人は東アジア人と南アジアの先住民(AASI)と同じ東ユーラシア人のソース集団に由来することが明らかになっている。[5] |
| https://en.wikipedia.org/wiki/Genetic_history_of_East_Asians |
|
リ ンク
文 献
そ の他の情報
Copyleft, CC, Mitzub'ixi Quq Chi'j, 1996-2099
☆
 ☆
☆