ハプロタイプ研究
haploid genotype studies
DNA molecule 1 differs from DNA molecule 2 at a single base-pair location (a C/A polymorphism).
DNA分子1はDNA分子2と1塩基対の位置で異なっている(C/A多型)。
ハプロタイプ研究
haploid genotype studies
DNA molecule 1 differs from DNA molecule 2 at a single base-pair location (a C/A polymorphism).
DNA分子1はDNA分子2と1塩基対の位置で異なっている(C/A多型)。
●EIC ネットでは「複数の対立遺伝子で、それぞれについてどちらの親から受け継い だ遺伝子かで分けたときに、片親由来の遺伝子の並びをハプロ タイプ(haploid genotype)と呼ぶ」とある。この解説はそれに続き次 のように文章を続けている。最後まで引用する:「染色体は、両親由来のものが2本1組で構成され、それぞれの遺伝子座の遺伝子(対立遺伝子)の組み合わせ により発現する形質が決まる。この対立遺伝子の組み合わせを遺伝子型と呼び、実際に発現する形質を表現型と呼ぶ。例えば、血液型のA型は、AAもしくは AOの組み合わせ(遺伝子型)があり、A型の表現型を示す。/親から子への遺伝は、染色体を最小単位とするため、ハプロタイプを得ることで、遺伝にかかわ るより完全なデータを得ることができる。例えば、ミトコンドリアDNAのハプロタイプ分析は、同一種内の地域集団分化に関する研究手法として用いられ、遺 伝的多様性保全のための重要な情報となっている。」
☆またGoogle
AIは次のようにハプロタイプを定義し、その特徴を述べる:「ハプロタイプ(haplotype)とは、遺伝子型(ジェノタイプ)の1本(単倍体)の染色体に注目した塩基配列の型です。
ハプロタイプの主な特徴は次のとおりです。
1)片親から受け継いだ遺伝子の並びを指します。
2)染色体は両親から2本1組で構成されており、それぞれの遺伝子座の遺伝子(対立遺伝子)の組み合わせにより発現する形質が決まります。
3)多くの場合、ハプロタイプはそのままの形で子供に受け継がれるため、タイピングの際に予測できます。
4)類似したハプロタイプを持つ集団のことを「ハプログループ」と呼び、ヒト祖先集団の誕生や移動を推定する研究に活用されています。
5)ミトコンドリアDNAのハプロタイプ分析は、同一種内の地域集団分化に関する研究手法として用いられ、遺伝的多様性保全のための重要な情報となっています。」(→なぜミトコンドリアハプロタイプの分析が「遺伝的多様性保全のための重要な情報」になるのか、このHPの提供者(池田)は十分理解しているとは言えないので、今後検討が必要)。
| A haplotype (haploid
genotype) is a group of alleles in an organism that are inherited
together from a single parent.[1][2] Many organisms contain genetic material (DNA) which is inherited from two parents. Normally these organisms have their DNA organized in two sets of pairwise similar chromosomes. The offspring gets one chromosome in each pair from each parent. A set of pairs of chromosomes is called diploid and a set of only one half of each pair is called haploid. The haploid genotype (haplotype) is a genotype that considers the singular chromosomes rather than the pairs of chromosomes. It can be all the chromosomes from one of the parents or a minor part of a chromosome, for example a sequence of 9000 base pairs or a small set of alleles. Specific contiguous parts of the chromosome are likely to be inherited together and not be split by chromosomal crossover, a phenomenon called genetic linkage.[3][4] As a result, identifying these statistical associations and a few alleles of a specific haplotype sequence can facilitate identifying all other such polymorphic sites that are nearby on the chromosome (imputation).[5] Such information is critical for investigating the genetics of common diseases; which in fact have been investigated in humans by the International HapMap Project.[6][7] Other parts of the genome are almost always haploid and do not undergo crossover: for example, human mitochondrial DNA is passed down through the maternal line and the Y chromosome is passed down the paternal line. In these cases, the entire sequence can be grouped into a simple evolutionary tree, with each branch founded by a unique-event polymorphism mutation (often, but not always, a single-nucleotide polymorphism (SNP)). Each clade under a branch, containing haplotypes with a single shared ancestor, is called a haplogroup.[8][9][10] |
ハプロタイプ(haploid
genotype)とは、生物の対立遺伝子のグループで、一つの親から一緒に受け継がれるものである[1][2]。 多くの生物は、2人の親から受け継いだ遺伝物質(DNA)を持っている。通常、これらの生物はDNAを2組の対になった染色体で構成している。子孫はそれ ぞれの親から1対ずつの染色体を受け継ぐ。対の染色体を2倍体といい、1対の染色体が半分ずつしかないものをハプロイドという。倍体の遺伝子型(ハプロタ イプ)は、染色体の対ではなく、単数の染色体を考慮した遺伝子型である。ハプロタイプは、両親の染色体のすべてであることもあれば、染色体のごく一部、例 えば9000塩基対の塩基配列や対立遺伝子の小さな集合であることもある。 染色体の特定の連続した部分は一緒に遺伝する可能性が高く、染色体のクロスオーバーによって分割されることはない。これは遺伝的連鎖と呼ばれる現象です [3][4]。その結果、特定のハプロタイプ配列の統計的関連と少数の対立遺伝子を特定することで、染色体上の近くにある他の多型部位をすべて特定するこ とが容易になる(インピュテーション)[5]。 例えば、ヒトのミトコンドリアDNAは母系で受け継がれ、Y染色体は父系で受け継がれる。このような場合、全塩基配列は単純な進化系統樹にまとめることが でき、各枝は一意的な多型変異(一塩基多型(SNP)であることが多いが、必ずしもそうではない)によって形成される。1つの枝の下にあるそれぞれのク レードは、単一の共有祖先を持つハプロタイプを含み、ハプログループと呼ばれる[8][9][10]。 |
| Haplotype resolution An organism's genotype may not define its haplotype uniquely. For example, consider a diploid organism and two bi-allelic loci (such as SNPs) on the same chromosome. Assume the first locus has alleles A or T and the second locus G or C. Both loci, then, have three possible genotypes: (AA, AT, and TT) and (GG, GC, and CC), respectively. For a given individual, there are nine possible configurations (haplotypes) at these two loci (shown in the Punnett square below). For individuals who are homozygous at one or both loci, the haplotypes are unambiguous - meaning that there is not any differentiation of haplotype T1T2 vs haplotype T2T1; where T1 and T2 are labeled to show that they are the same locus, but labeled as such to show it does not matter which order you consider them in, the end result is two T loci. For individuals heterozygous at both loci, the gametic phase is ambiguous - in these cases, an observer does not know which haplotype the individual has, e.g., TA vs AT. 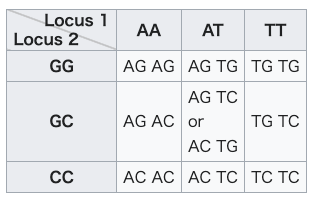 The only unequivocal method of resolving phase ambiguity is by sequencing. However, it is possible to estimate the probability of a particular haplotype when phase is ambiguous using a sample of individuals. Given the genotypes for a number of individuals, the haplotypes can be inferred by haplotype resolution or haplotype phasing techniques. These methods work by applying the observation that certain haplotypes are common in certain genomic regions. Therefore, given a set of possible haplotype resolutions, these methods choose those that use fewer different haplotypes overall. The specifics of these methods vary - some are based on combinatorial approaches (e.g., parsimony), whereas others use likelihood functions based on different models and assumptions such as the Hardy–Weinberg principle, the coalescent theory model, or perfect phylogeny. The parameters in these models are then estimated using algorithms such as the expectation-maximization algorithm (EM), Markov chain Monte Carlo (MCMC), or hidden Markov models (HMM). Microfluidic whole genome haplotyping is a technique for the physical separation of individual chromosomes from a metaphase cell followed by direct resolution of the haplotype for each allele. Gametic phase In genetics, a gametic phase represents the original allelic combinations that a diploid individual inherits from both parents.[11] It is therefore a particular association of alleles at different loci on the same chromosome. Gametic phase is influenced by genetic linkage.[12] |
ハプロタイプの決定 生物の遺伝子型がハプロタイプを一意に定義するとは限らない。例えば、2倍体の生物で、同じ染色体上に2つの2対立遺伝子座(SNPなど)があるとする。 最初の遺伝子座は対立遺伝子がAまたはTで、2番目の遺伝子座はGまたはCであると仮定する。ある個体について、これら2つの遺伝子座には9つの可能な構 成(ハプロタイプ)がある(下のパネットスクエアに示す)。片方または両方の遺伝子座でホモ接合の個体では、ハプロタイプは一義的である。つまり、ハプロ タイプT1T2とハプロタイプT2T1は区別されない。T1とT2は同じ遺伝子座であることを示すためにラベルが貼られているが、どの順番で考えても最終 的には2つのT遺伝子座になることを示すためにそのようにラベルが貼られている。両方の遺伝子座でヘテロ接合の個体では、配偶相はあいまいである。このよ うな場合、観察者はその個体がどちらのハプロタイプを持っているのかわからない。  相のあいまいさを解決する唯一の明確な方法は、配列決定である。しかし、相があいまいな場合、個体のサンプルを使って特定のハプロタイプの確率を推定する ことは可能である。 多数の個体の遺伝子型が与えられた場合、ハプロタイプはハプロタイプ分解法またはハプロタイプ位相決定法によって推定することができる。これらの方法は、 特定のハプロタイプが特定のゲノム領域で一般的であるという観察を応用することで機能する。したがって、可能なハプロタイプ解像度のセットが与えられた場 合、これらの方法は、全体としてより少ない異なるハプロタイプを使用するものを選択する。これらの手法の具体的な内容はさまざまで、組み合わせアプローチ (例えばパーシモン)に基づくものもあれば、ハーディー・ワインベルグの原理、合体理論モデル、完全系統などの異なるモデルや仮定に基づく尤度関数を用い るものもある。これらのモデルのパラメータは、期待値最大化アルゴリズム(EM)、マルコフ連鎖モンテカルロ(MCMC)、隠れマルコフモデル(HMM) などのアルゴリズムを用いて推定される。 マイクロ流体全ゲノムハプロタイピングは、メタフェース細胞から個々の染色体を物理的に分離し、各アレルのハプロタイプを直接決定する技術である。 配偶相 遺伝学では、配偶相は2倍体個体が両親から受け継ぐ対立遺伝子の組み合わせを表す[11]。配偶相は遺伝的連鎖の影響を受ける[12]。 |
| Y-DNA haplotypes from
genealogical DNA tests Main article: Genealogical DNA test Unlike other chromosomes, Y chromosomes generally do not come in pairs. Every human male (excepting those with XYY syndrome) has only one copy of that chromosome. This means that there is not any chance variation of which copy is inherited, and also (for most of the chromosome) not any shuffling between copies by recombination; so, unlike autosomal haplotypes, there is effectively not any randomisation of the Y-chromosome haplotype between generations. A human male should largely share the same Y chromosome as his father, give or take a few mutations; thus Y chromosomes tend to pass largely intact from father to son, with a small but accumulating number of mutations that can serve to differentiate male lineages. In particular, the Y-DNA represented as the numbered results of a Y-DNA genealogical DNA test should match, except for mutations. UEP results (SNP results) Unique-event polymorphisms (UEPs) such as SNPs represent haplogroups. STRs represent haplotypes. The results that comprise the full Y-DNA haplotype from the Y chromosome DNA test can be divided into two parts: the results for UEPs, sometimes loosely called the SNP results as most UEPs are single-nucleotide polymorphisms, and the results for microsatellite short tandem repeat sequences (Y-STRs). The UEP results represent the inheritance of events it is believed can be assumed to have happened only once in all human history. These can be used to identify the individual's Y-DNA haplogroup, his place in the "family tree" of the whole of humanity. Different Y-DNA haplogroups identify genetic populations that are often distinctly associated with particular geographic regions; their appearance in more recent populations located in different regions represents the migrations tens of thousands of years ago of the direct patrilineal ancestors of current individuals. Y-STR haplotypes Genetic results also include the Y-STR haplotype, the set of results from the Y-STR markers tested. Unlike the UEPs, the Y-STRs mutate much more easily, which allows them to be used to distinguish recent genealogy. But it also means that, rather than the population of descendants of a genetic event all sharing the same result, the Y-STR haplotypes are likely to have spread apart, to form a cluster of more or less similar results. Typically, this cluster will have a definite most probable center, the modal haplotype (presumably similar to the haplotype of the original founding event), and also a haplotype diversity — the degree to which it has become spread out. The further in the past the defining event occurred, and the more that subsequent population growth occurred early, the greater the haplotype diversity will be for a particular number of descendants. However, if the haplotype diversity is smaller for a particular number of descendants, this may indicate a more recent common ancestor, or a recent population expansion. It is important to note that, unlike for UEPs, two individuals with a similar Y-STR haplotype may not necessarily share a similar ancestry. Y-STR events are not unique. Instead, the clusters of Y-STR haplotype results inherited from different events and different histories tend to overlap. In most cases, it is a long time since the haplogroups' defining events, so typically the cluster of Y-STR haplotype results associated with descendants of that event has become rather broad. These results will tend to significantly overlap the (similarly broad) clusters of Y-STR haplotypes associated with other haplogroups. This makes it impossible for researchers to predict with absolute certainty to which Y-DNA haplogroup a Y-STR haplotype would point. If the UEPs are not tested, the Y-STRs may be used only to predict probabilities for haplogroup ancestry, but not certainties. A similar scenario exists in trying to evaluate whether shared surnames indicate shared genetic ancestry. A cluster of similar Y-STR haplotypes may indicate a shared common ancestor, with an identifiable modal haplotype, but only if the cluster is sufficiently distinct from what may have happened by chance from different individuals who historically adopted the same name independently. Many names were adopted from common occupations, for instance, or were associated with habitation of particular sites. More extensive haplotype typing is needed to establish genetic genealogy. Commercial DNA-testing companies now offer their customers testing of more numerous sets of markers to improve definition of their genetic ancestry. The number of sets of markers tested has increased from 12 during the early years to 111 more recently. Establishing plausible relatedness between different surnames data-mined from a database is significantly more difficult. The researcher must establish that the very nearest member of the population in question, chosen purposely from the population for that reason, would be unlikely to match by accident. This is more than establishing that a randomly selected member of the population is unlikely to have such a close match by accident. Because of the difficulty, establishing relatedness between different surnames as in such a scenario is likely to be impossible, except in special cases where there is specific information to drastically limit the size of the population of candidates under consideration. |
系図DNA検査によるY-DNAハプロタイプ 主な記事 系図DNA検査 他の染色体とは異なり、Y染色体は通常ペアで存在しません。すべてのヒト男性(XYY症候群の人を除く)は、Y染色体のコピーを1本しか持っていません。 つまり、どのコピーが受け継がれるかは偶然ではなく、また(染色体の大部分では)組換えによるコピー間のシャッフルもありません。従って、常染色体のハプ ロタイプとは異なり、Y染色体のハプロタイプは世代間でランダムに変化することは事実上ありません。従って、Y染色体は父から子へとほぼそのまま受け継が れる傾向があるが、突然変異の数は少ない。特に、Y-DNA系図DNA鑑定の結果番号で表されるY-DNAは、突然変異を除いて一致するはずです。 UEP結果(SNP結果) SNPなどの特異事象多型(UEP)はハプログループを表します。STRはハプロタイプを表します。Y染色体DNA検査によるY-DNAハプロタイプの結 果は、UEPの結果(ほとんどのUEPは一塩基多型であるため、SNPの結果と呼ばれることもあります)と、マイクロサテライト・ショートタンデムリピー ト配列(Y-STR)の結果の2つに分けられます。 UEPの結果は、全人類の歴史の中で一度だけ起こったと考えられる出来事の遺伝を表している。これらの結果は、個人のY-DNAハプログループ、つまり人 類全体の「家系図」における位置を特定するために使用することができる。異なるY-DNAハプログループは、しばしば特定の地理的地域と明確に関連する遺 伝的集団を特定する。異なる地域に位置する、より最近の集団におけるそれらの出現は、現在の個人の直接の父系祖先が数万年前に移動したことを表している。 Y-STRハプロタイプ 遺伝学的結果にはY-STRハプロタイプも含まれ、これは検査したY-STRマーカーの結果の集合である。 UEPとは異なり、Y-STRは変異しやすいため、最近の系譜を区別するために使用することができる。しかし、それはまた、ある遺伝的事象の子孫の集団が すべて同じ結果を共有するのではなく、Y-STRハプロタイプがばらばらに広がって、多かれ少なかれ似たような結果のクラスターを形成している可能性が高 いことを意味します。一般的に、このクラスターは、最も可能性の高い中心であるモーダルハプロタイプ(おそらく最初の発見事象のハプロタイプに似ている) と、ハプロタイプの多様性(どの程度広がっているか)を持っています。ハプロタイプ多様性とは、ハプロタイプがどの程度拡散しているかということである。 決定的な出来事が過去に起これば起こるほど、またその後の人口増加が早期に起これば起こるほど、特定の数の子孫のハプロタイプ多様性は大きくなる。しか し、特定の数の子孫に対してハプロタイプの多様性が小さい場合、これはより最近の共通祖先、あるいは最近の集団拡大を示している可能性がある。 UEPの場合とは異なり、同じY-STRハプロタイプを持つ2人の個体が必ずしも同じ祖先を持つとは限らないことに注意することが重要である。Y-STR 事象は一意ではない。その代わり、異なる事象や異なる歴史から受け継いだY-STRハプロタイプの結果のクラスターは重なる傾向がある。 ほとんどの場合、ハプログループを定義する出来事から長い時間が経過しているため、通常、その出来事の子孫に関連するY-STRハプロタイプの結果のクラ スターは、かなり幅広くなっています。これらの結果は、他のハプログループに関連する(同様に広い)Y-STRハプロタイプのクラスターとかなり重なる傾 向がある。このため、研究者がY-STRハプロタイプがどのY-DNAハプログループを指すかを絶対的に確実に予測することは不可能である。UEPを検査 しない場合、Y-STRはハプログループの祖先の確率を予測するために使用されるだけで、確実なものではありません。 同じようなシナリオは、共有姓が遺伝的祖先の共有を示すかどうかを評価しようとする場合にも存在する。類似したY-STRハプロタイプのクラスターは、識 別可能な様式ハプロタイプを持つ共通の祖先が共有されていることを示すかもしれないが、そのクラスターが、歴史的に同じ名前を独自に採用した異なる個人か ら偶然に起こった可能性のあるものとは十分に異なる場合に限られる。例えば、多くの名前は共通の職業から採用されたものであったり、特定の場所での居住に 関連したものであったりする。遺伝的系図を確立するためには、より広範なハプロタイプのタイピングが必要である。市販のDNA検査会社は現在、遺伝的先祖 の定義を改善するために、より多くのマーカーセットの検査を顧客に提供している。検査されるマーカーの数は、初期の12セットから最近では111セットに 増えている。 データベースからデータマイニングされた異なる姓の間のもっともらしい血縁関係を確立することは、かなり困難である。研究者は、問題となっている集団の最 も近いメンバーが、そのために意図的に集団から選ばれたものであり、偶然に一致する可能性が低いことを立証しなければならない。これは、母集団から無作為 に選ばれたメンバーが、偶然にそのような近い一致をする可能性が低いことを立証する以上に難しいことである。その難しさゆえに、このようなシナリオのよう に異なる姓の間の血縁関係を確立することは、候補者の母集団の大きさを大幅に制限するような特別な情報がある場合を除き、おそらく不可能であろう。 |
| Diversity Haplotype diversity is a measure of the uniqueness of a particular haplotype in a given population. The haplotype diversity (H) is computed as:[13] 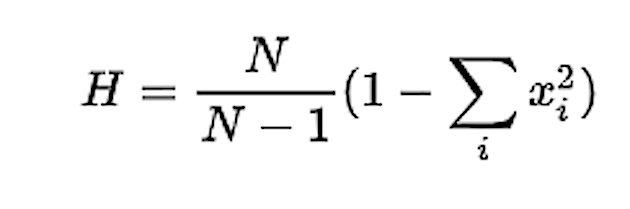 where Xi is the (relative) haplotype frequency of each haplotype in the sample and N is the sample size. Haplotype diversity is given for each sample. |
多様性 ハプロタイプの多様性は、ある集団における特定のハプロタイプの独自性を示す尺度である。ハプロタイプ多様性(H)は次のように計算される[13]。 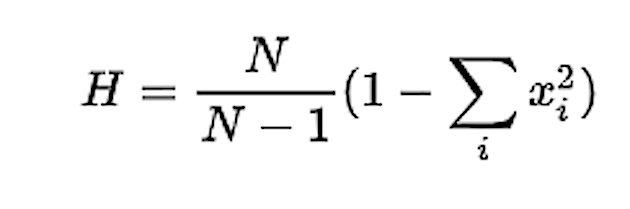 ここで、Xiは標本中の各ハプロタイプの(相対)ハプロタイプ頻度、Nは標本サイズである。ハプロタイプの多様性は各サンプルについて与えられる。 |
| https://en.wikipedia.org/wiki/Haplotype |
|
| ハプロタイプ(haplotype)とは、"haploid
genotype"(半数体の遺伝子型)の略。単倍型とも訳される。 概要 ハプロタイプは、生物がもっている単一の染色体上の遺伝的な構成(具体的にはDNA配列)のことである。二倍体生物の場合、ハプロタイプは各遺伝子座位に ある対立遺伝子のいずれか一方の組合せをいう。 またゲノム全体に対して(複数の染色体にまたがって)いうこともあるが、この場合には特にいずれかの片親に由来する遺伝子の組合せを指す。通常、母系のミ トコンドリアと、父系のY染色体が対象となる。 さらに現在は限定的な意味として、同一染色体上で統計学的に見て関連のある、つまり遺伝的に連鎖している多型(一塩基多型[SNP]など)の組合せをいう ことが多い。このような組合せがわかれば、ある範囲内について、少数の対立遺伝子を同定することで他の多型座位も決めることができる。このような情報は疾 病の遺伝的な要因を 調べるのに特に有用であり、現在これらを収集し解析する国際的なプロジェクト、International HapMap Projectが進められた。 補足 遺伝子型とハプロタイプは別の概念であり、個体の遺伝子型からその個体のハプロタイプを一意的に決めることはできない。 例として2つの遺伝子座位を考え、それぞれに2種の対立遺伝子(第1がAまたはa、第2がBまたはb)があるとしよう。ある個体の遺伝子型がAaBbであ ることがわかったら、同じ染色体にどのペアがあるかによってハプロタイプのセットには2つの可能性がある:  この場合、どの特定のハプロタイプのセットが個体に現れているか(つまりどの対立遺伝子が同じ染色体上にあるか)を決定するにはさらなる情報が必要とな る。 多数の個体に対して遺伝子型がわかれば、これらに共通のハプロタイプがあると考えてハプロタイプを推定できる。可能な一組のハプロタイプがわかれば、次に なるべく少数のハプロタイプで説明できる方法を選択する。この方法には様々なものがあり、例えば最大節約法や、尤度関数とそれを計算するアルゴリズム(期 待値最大化アルゴリズムExpectation-maximization algorithm(EM)やマルコフ連鎖モンテカルロ法Markov Chain Monte Carlo(MCMC))を用いる方法がある。 ハプロタイプの応用 現在最も期待されているのは、既述のように疾病の遺伝的要因の解析への応用である。 ハプロタイプは親から子へ引き継がれるものであるから家系調査にも応用できる。特に男性のY染色体のハプロタイプは父系の調査に用いられる。Y染色体のハ プロタイプは他のハプロタイプと違ってペアをなさないから、他の染色体との乗換えが起こらずに父から息子へと伝えられる。同じ姓をもつ様々な子孫を調べた 場合、その姓に対する標準的なY染色体ハプロタイプが見られることもある。これはその姓を名乗った最も古い共通祖先のハプロタイプである可能性が高い。 逆にミトコンドリアDNAのハプロタイプは母系を推定するのに用いられている。 ハプロタイプは人類などの集団の比較にも用いられる。ハプロタイプには多様性があり、近い集団では似ているが遠い集団では大きく異なる。ハプロタイプを大 きくまとめたものをハプログループといい、これは遺伝的集団を示す指標として用いられ、また地理的なまとまりを見せる場合が多いので、人類の移住の歴史を 推定するのにも用いられる。 ハプログループ よく似たハプロタイプの集団を、ハプログループという。これは単倍群とも訳される。ハプロタイプは一つでは意味がなく、たくさんのハプロタイプを統計的に 処理した場合のみ、意味を持つ。そのとき、似たハプロタイプからなるハプログループが研究対象となる。いくつかのハプログループの系統関係を調べること で、生物(特に人間)の系統を知ることができる。ミトコンドリア・イブやY染色体ハプログループを参照。 |
●
| In human genetics, Haplotype 35,
also called ht35 or the Armenian Modal Haplotype, is a Y chromosome
haplotype of Y-STR microsatellite variations, associated with the
Haplogroup R1b. It is characterized by DYS393=12 (as opposed to the
Atlantic Modal Haplotype, another R1b haplotype, which is characterized
by DYS393=13). The members of this haplotype are found in high numbers
in Anatolia and Armenia, with smaller numbers throughout Central Asia,
the Middle East, the Balkans, the Caucus Mountains, and in Jewish
populations. They are also present in Britain in areas that were found
to have a high concentration of Haplogroup J, suggesting they arrived
together, perhaps through Roman soldiers.[1] https://en.wikipedia.org/wiki/Haplotype_35 |
人
類遺伝学において、ハプロタイプ35(ht35またはアルメニア・モーダル・ハプロタイプとも呼ばれる)は、ハプログループR1bに関連するY-STRマ
イクロサテライト変異のY染色体ハプロタイプである。このハプロタイプの特徴はDYS393=12である(DYS393=13であるもう1つのR1bハプ
ロタイプ、アトランティック・モーダル・ハプロタイプとは対照的である)。このハプロタイプのメンバーはアナトリアとアルメニアに多く、中央アジア、中
東、バルカン半島、コーカス山脈、ユダヤ人の集団には少ないが見られる。このハプロタイプはイギリスでもハプログループJが集中している地域に存在してお
り、おそらくローマ兵を通じて一緒にやってきたことを示唆している[1]。 |
用語解説
用語解説
ハプロタイプ(haplotype)とは、"haploid
genotype"(半数体の遺伝子型)の省略語である。
ハプログループ(haplogroup):単一のDNA一塩基多型 (SNP) ——ある生物種集団のゲノム塩基配列中に一塩基が変異した多様性——変異がある共通祖先をもつよく似たハプロタイプ(haploid genotype=半数体の遺伝子型)の集団のこと。
A haplotype is a group of alleles -- a variant form of a given gene -- in an organism that are inherited together from a single parent, and a haplogroup (haploid from the Greek: ἁπλούς, haploûs, "onefold, simple" and English: group) is a group of similar haplotypes that share a common ancestor with a single-nucleotide polymorphism mutation.
ハプログループY=Y染色体ハプログループ(Human Y-chromosome DNA haplogroup)父系で遺伝するY染色体のハプログループ(=ハプロタイプの集団)のこと。「言語学上 の区分に近いが、外見上の人種区分とは違うパターンが少なからずある。これは遺伝子の系統と集団の系統が異なる(incomplete lineage sorting)による」Y 染色体ハプログループ)
DNA型鑑定(DNA profiling):デオキシリボ核酸 (DNA)
の多型部位を検査することで個人や集団を識別するために行う鑑定方法。
二 重構造モデル(Dual structure model for the population history of the Japanese): 埴原和郎は、沖縄人およびアイヌを含む日本人集団の形成史を単一の仮説で説明するモデルを提唱したが、このことをいう。埴原によると、「日本列島の最初の 居住者は後期旧石器時代に移動してきた東南アジア系の集団で, 縄文人はその子孫である。弥生時代になって第2の移動の波が北アジアから押し寄せたため, これら2系統の集団は列島内で徐々に混血した。この混血の過程は現在も続いており, 日本人集団の二重構造性は今もなお解消されていない。したがって身体・文化の両面にみられる日本の地域性-たとえば東西日本の差など-は, 混血または文化の混合の程度が地域によって異なるために生じたと説明することができる。またこのモデルは, 日本人の形質・文化にみられるさまざまな現象を説明するのみならず, イヌやハツカネズミなど, 人間以外の動物を対象とする研究結果にも適合する。同時に, このモデルによって日本の本土, 沖縄およびアイヌ系各集団の系統関係も矛盾なく説明することができる」という(→「二重構造モデル」)。
ミトコンドリア(mitochondrion、
複数形:
mitochondria)は真核生物の細胞小器官。「ミトコンドリア中にはDNAが存在しており、ここに細胞核のものとは異なる独自の遺伝情報を持って
いる。通常はGC含量が低く(20-40%)、基本的なゲノムのサイズは数十kb程度のDNAであり、電子伝達系に関わるタンパク質、リボソームRNAや
tRNAなど数十種類の遺伝子がある」ミトコンドリア)
●Y染色体ハプログループの系統と分岐年代。出アフリカ組(D1, C, FT)の下位系統の放散は約5万年前に集中している。PからRとQへの分岐は例外的に約3万5000年前に起きている。
「Y染色体の最新共通祖先 (Y-MRCA、非公式にはY 染色体 Adamとして知られている) は、現在生きているすべての人間が父系で子孫を残した最新共通祖先 (MRCA )である。 Y染色体を持つアダムは、およそ 236,000年前にアフリカに住んでいたと推定されている。他のボトルネックを調べると、ほとんどのユーラシア人男性 (アフリカ以外の人口からの男性) は、69,000年前にアフリカに住んでいた男性 ( Haplogroup_CT ) の子孫である。 他の主要なボトルネックは約5万年前と5千年前に発生し、その後、ほとんどのユーラシア人の祖先は、5万年前に生きていた4 人の祖先に遡ることができた。彼らはアフリカ人の子孫だった (E-M168)。言語学上の語族の分布と相関性が高い(父系言語仮説[1])が、外見上の人種区分とは違うパターンが少なからずある(これは遺伝子の系 統と集団の系統が異なる不完全遺伝子系統仕分けによるものである)」(出典「Y染色体ハプロ グループ」)
父系言語仮説[1] van Driem, George. 2018. "The East Asian linguistic phylum: A reconstruction based on language and genes Archived 2021-01-10 at the Wayback Machine.", Journal of the Asiatic Society, LX (4): 1-38.[2018i.pdf]with password
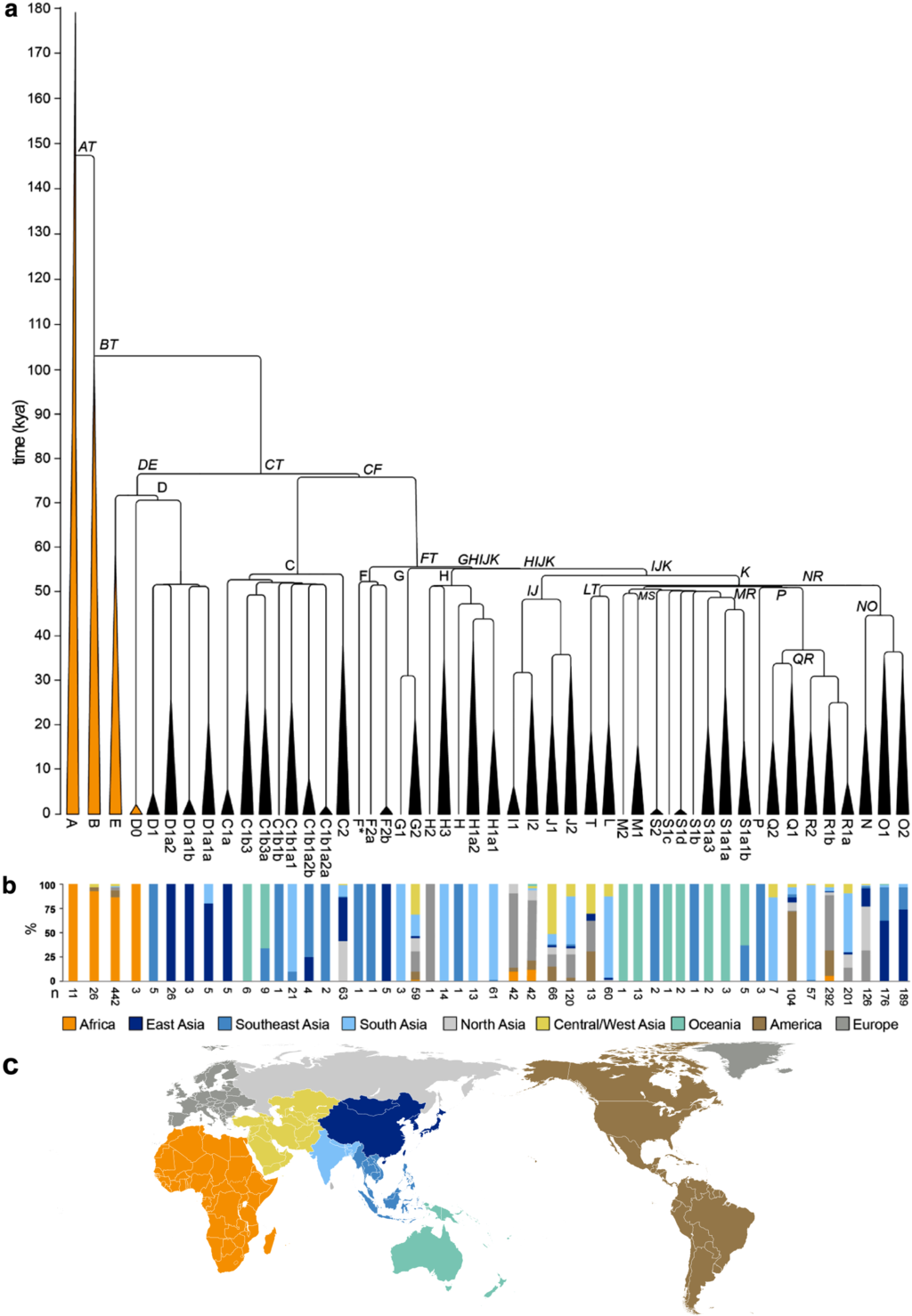
| Y染色体ハプログループ(Yせんしょくたいハ
プログループ)とは、男性で遺伝するY染色体のハプログループ(=ハプロタイプの集団)のことである。 概略 Y染色体ハプログループの系統と分岐年代。出アフリカ組(D1, C, FT)の下位系統の放散は約5万年前に集中している。PからRとQへの分岐は例外的に約3万5000年前に起きている。 Y染色体の最新共通祖先 (Y-MRCA、非公式にはY 染色体 Adamとして知られている) は、現在生きているすべての人間が父系で子孫を残した最新共通祖先 (MRCA )である。 Y染色体を持つアダムは、およそ 236,000年前にアフリカに住んでいたと推定されている。他のボトルネックを調べると、ほとんどのユーラシア人男性 (アフリカ以外の人口からの男性) は、69,000年前にアフリカに住んでいた男性 ( Haplogroup_CT ) の子孫である。 他の主要なボトルネックは約5万年前と5千年前に発生し、その後、ほとんどのユーラシア人の祖先は、5万年前に生きていた4 人の祖先に遡ることができた。彼らはアフリカ人の子孫だった (E-M168)。言語学上の語族の分布と相関性が高い(父系言語仮説[1])が、外見上の人種区分とは違うパターンが少なからずある(これは遺伝子の系 統と集団の系統が異なる不完全遺伝子系統仕分けによるものである)。 |
|
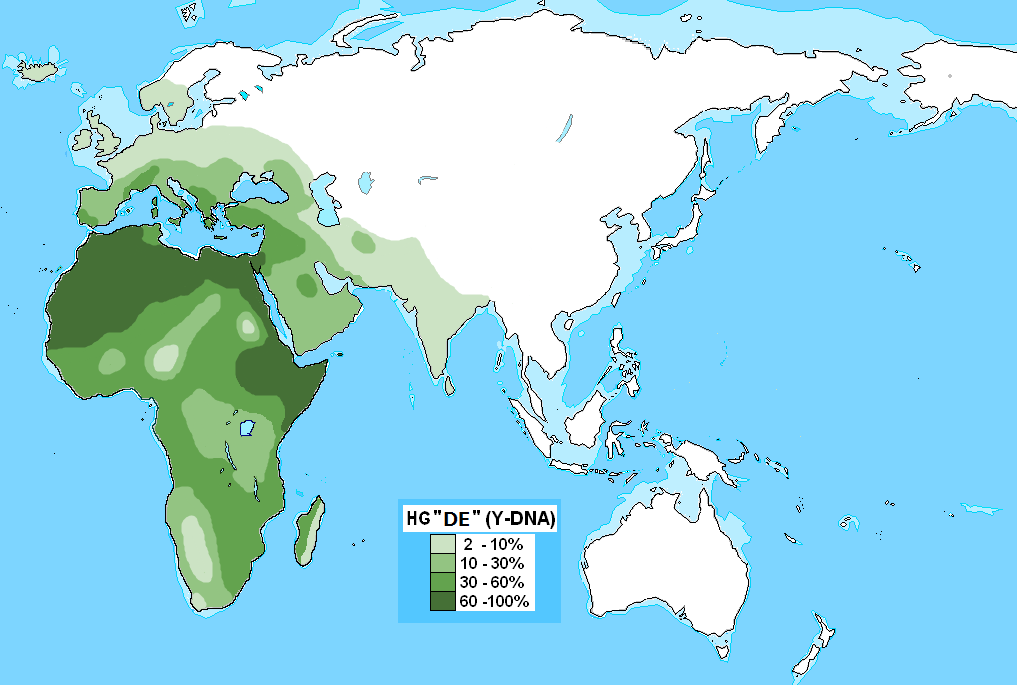 |
ハ
プログループE (Y染色体)(ハプログループE
(Yせんしょくたい)とは、分子人類学で用いられる、人類のY染色体ハプログループの分類のうち、ハプログループDE
(Y染色体)のサブクレード(細分岐)の一つで、「L339, L614, M40/SRY4064/SRY8299, M96, P29,
P150, P152, P154, P155, P156, P162, P168, P169, P170, P171, P172, P173,
P174, P175,
P176」の変異で定義づけられる系統である。約7.3万年前[1]に東アフリカ[2][3][1]または西アジア[4]で誕生した。https://x.gd/EkSKE |
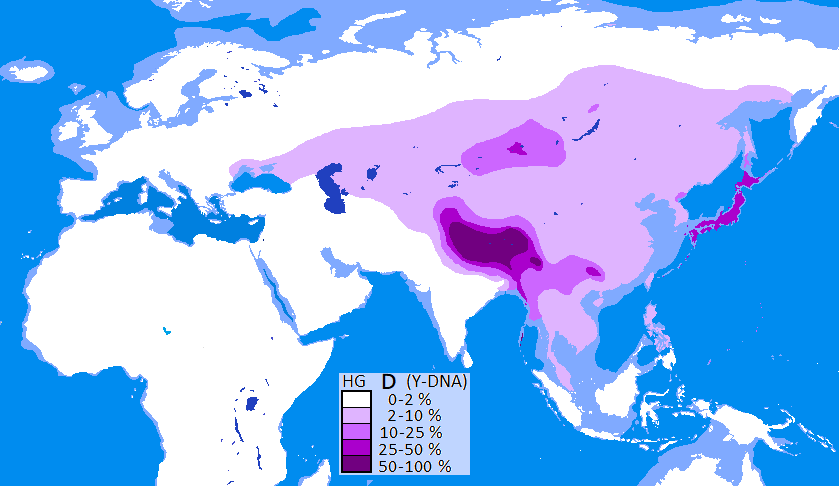 |
ハ
プログループD (Y染色体)(ハプログループD (Yせんしょくたい)、英: Haplogroup D
(Y-DNA))とは、分子人類学で用いられる、人類のY染色体ハプログループの分類で、YAPと呼ばれる変異の型を持つもののうちのCTS3946に代
表される分岐指標を持つ集団の系統。 分布 現在このハプログループDは、日本列島・南西諸島やアンダマン諸島、チベット高原で高頻度に観察されるほかはアジア、アフリカの極めて限られた地域で散発 的にしか見つかっていない。 チベットではD1a1-Z27276、日本ではD1a2a⁻M55、アンダマン諸島ではD1a2b-Y34537[4] が高頻度である。 これらのハプログループは、同じハプログループDに属していてもサブグループが異なるため、分岐してから5万3000年以上の年月を経ている[5]。 ハプログループDは、現在の中国、朝鮮、東南アジアにおいて多数派的なハプログループO系統や、その他E系統以外のユーラシア系統(C,I,J,N,Rな ど)とは分岐から7万年以上の隔たりがあり、非常に孤立的な系統となっている。 D系統は東アジアにおける最古層のタイプと想定できるが[6]、一つの説として東アジア及び東南アジアにO系統が広く流入した為、島国日本や山岳チベット にのみD系統が残ったと考えられている。 なお、同じくハプログループDEから分かれたハプログループEは、アフリカ大陸で高頻度、中東や地中海地域で中~低頻度に見られる。 またDEの子型でD系統にもE系統にも属さないDE*がチベット人でごくわずかに発見されている[7]。 起源 最近の研究 (Haber et al. 2019)[1] で、従来DEの子型でD系統にもE系統にも属さないパラグループDE*(どちらの系統に近いか未詳)とされていたナイジェリア人の3サンプル[8] が、ハプログループEよりもハプログループDに近縁であることが判明した。このサンプルは、ハプログループEの持たないSNPを、ハプログループDと7つ 共有しており、「D2」としてハプログループDに組み入れられた。 この発見により従来ハプログループD(M174の変異で定義)はハプログループD1に名称が変更され、新たに発見されたD2(D-A5580.2)とハプ ログループD1(M174)を併せ、ハプログループDはCT3946の変異によって定義されるグループへと拡大された。なおこのD2は、西アジア(サウジ アラビア、シリア)でも見つかっている[9]。 このようにハプログループD2がアフリカから見つかったことから、ハプログループD(CT3946)はアフリカで既に誕生していたと推定されている [1]。 従って、ハプログループDは、今より約7.3万年前にアフリカ[1] にてハプログループDEから発生、下位系統のハプログループD1のみが出アフリカを果たし、その後内陸ルートを通って東アジアへ向かったことになる [10]。(D2発見以前は、ハプログループDEとDがアジアで発祥したという異説[11] もあった。) なお、パラグループDE*はチベット人[6] や他の西アフリカのサンプル[12] でも検出されているが、これらについて詳細な分析は成されていない。 https://x.gd/iVxaP |
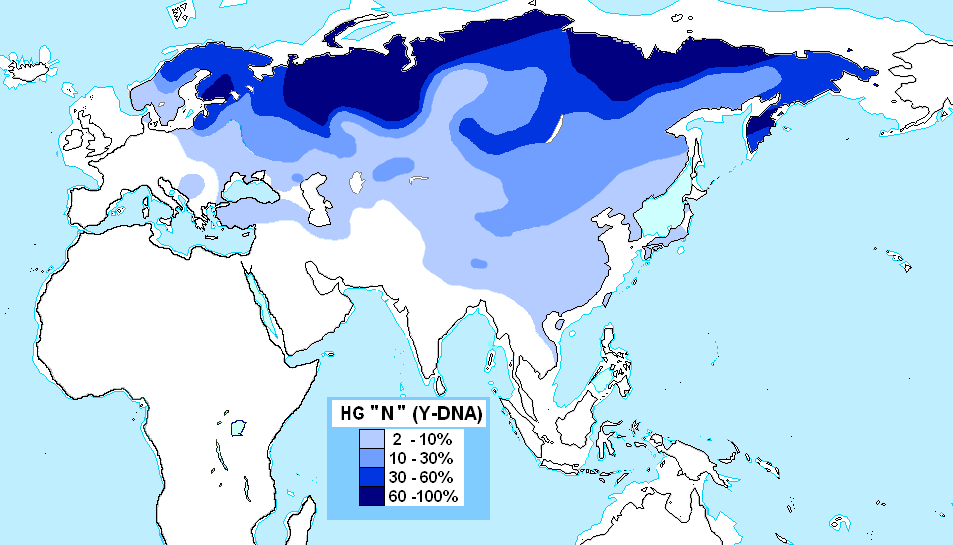 |
ハ
プログループN (Y染色体)(ハプログループN (Yせんしょくたい)、英: Haplogroup N
(Y-DNA))とは分子人類学において人類の父系を示すY染色体ハプログループ(型集団)の分類で、「M231」以下の系統に位置すると定義されるもの
である[5]。 起源・分布 Y染色体のハプログループNは、ユーラシア北部出身の男性に最もよく見られる。また、バルカン半島、中央アジア、東アジア、東南アジアの一部を含む他の地 域に自生する個体群では、より低い頻度で観察されている。 現存のY染色体ハプログループの中で最も近縁のハプログループであるハプログループOとはKarmin et al. (2015)によれば41,900年(95% CI 40,175年~43,591年)前[1]、Poznik et al. (2016)によれば44,700年あるいは38,300年前[2]、YFull (2017)によれば36,800年 (95% CI 34,300年~39,300年)前[3]に分岐をしたと推定されている。ハプログループNに属す現存のY染色体は20,000年前~25,000年前 [4]に東アジアにおいて分岐をし始めたと推定されており、ユーラシア北部、さらにはシベリアを横断して北欧まで分布を広げた[6]。観察頻度はネネツ人 に97%、 ガナサン人に92%、ヤクート人に88%[7]、 フィン人に63%[8]、エヴェンキ族に20-60%[9]、チュクチ人に58%[10]、サーミ人に47%[11]、エストニア人に41%[8]、ユカ ギール人に31%[12]、ロシア人に20%[13]などである。ウラル語族との関連が想定される。[独自研究?]フィン・ウゴル系にN1a1、サモエー ド系にN1a2が多い。 遼河文明の遺跡人骨からもN1が60%以上の高頻度で見つかっており[14][15]、かつては東アジア北部においても支配的であったと想定されるが、現 在においては概ね10%程度の低頻度となっている。 ハプログループ N-P43は北サモエド人、オブ・ウグリック語話者、北ハカス人の間で非常に頻繁に発見されており、他のウラル語話者、テュルク人、モンゴル人、ツングー ス人の間でも低頻度から中程度の頻度で観察されている。 https://x.gd/aBJG9 |
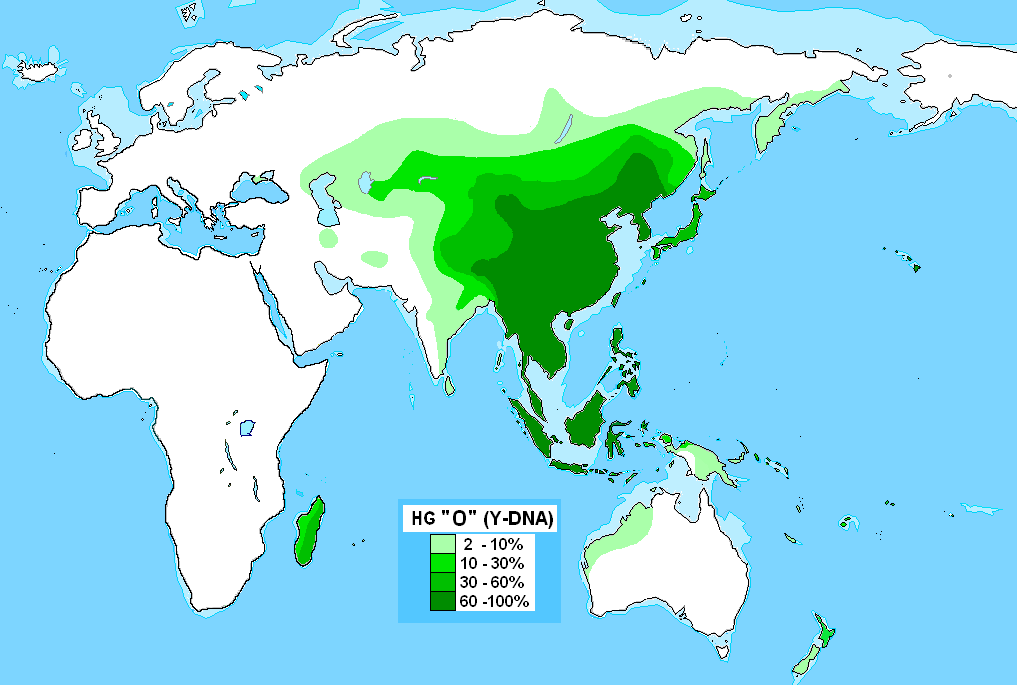 |
ハ
プログループO (Y染色体)(ハプログループO (Yせんしょくたい)、英: Haplogroup O
(Y-DNA))とは分子人類学において人類の父系を示すY染色体ハプログループ(型集団)の分類で、ハプログループNOの子型で「M175」以下の系統
に位置すると定義されるものである。東アジア及び東南アジアで最も一般的に見られる系統であり、西ユーラシア系のハプログループRと並んで現代人類におい
て最も帰属人口の多いY染色体であると考えられている。 概要 Y染色体のハプログループOは、おおよそ4万年前[注 1]にハプログループNと血筋が分かれた。 現存のハプログループO(即ちO1-F265とO2-M122)の最も近い共通祖先は、約3万年~3.5万年前[注 2]に生きたとされる。 華北平原や長江流域などの豊かな地域を中心に多岐に渡るサブグループを産み出し、これらの地域から東アジアや東南アジアからポリネシアに至るまで広範囲に 拡散したと考えられている[7]。 「大渓文化」の遺跡人骨から検出されたY染色体ハプログループは、現在は中国南部から東南アジア北部の山岳地帯に分布する「モン・ミエン語族(ミャオ・ヤ オ語族)」の民族に多い「O2a2a1a2 M7(旧O3系)」が主体(5/7)で、「O2*(旧O3*)」と「O1b1a1a M95(旧O2a系)」がそれぞれその一部(1/7)であった。 「大渓文化」は「屈家嶺文化」へ継承された。 大きくO1とO2に大別される[8]。約3万年前頃までには、O1a-M119, O1b1-K18, O1b2-M176, O2a-M324, O2b-F742 という五つの古い系統に分かれていたと推定されている[1][9][4]。 https://x.gd/jFEWv |
 |
ハ
プログループC-M217 (Y染色体)(ハプログループC-M217 Yせんしょくたい、英: Haplogroup C-M217
(Y-DNA))、系統名称ハプログループC2とは分子人類学において人類の父系を示すY染色体ハプログループ(型集団)の分類で、ハプログループCの下
位枝に属し、「M217, P44, PK2」によって定義されるグループである。かつてはハプログループC3と呼ばれていた。 約50,865年前[1]にC1-F3393/Z1426と共通の祖先から派生して、現存の全てのC-M217の最も近い共通祖先は約35,383年前 [1]にユーラシア大陸北東部で生まれたと考えられる。民族の総人口に占める割合としては、ユーラシアではツングース系民族、モンゴル系民族、カザフ、ハ ザーラ、コリャーク、イテリメン、ユカギール、ニヴフに多く見られ、アメリカ大陸ではナデネ語族話者に比較的多く見られる。 日本に於けるC2-M217の分布の地域差がかなり大きく、少ない所では全く観察されないサンプル(例えば東京52人中0人[2]、沖縄県立八重山高等学 校及び沖縄県立八重山商工高等学校の男子生徒49人中0人[3]、沖縄45人中0人[4]、青森26人中0人[4])もあれば、多い所では7.8%ほど観 察されるサンプルもある(佐賀県立致遠館高等学校の男子生徒129人中10人[3]、福岡市の成人男性102人中8人[5])。[4][6][7] サンプル数が少ないが、日本国内に於いて一番高い比率でC-M217に属すY-DNAが検出されているのはアイヌのサンプルである(16人中2人 = 12.5%[8]、4人中1人 = 25%[4])。日本国内でアイヌに次いでC2-M217の比率が多いのは概ね九州北部(大分県・福岡県・佐賀県・長崎県)であるが[5][3][9]、 韓国の研究チームが検査した茨城県50人のサンプルではC-M217に属す男性が7人(14%)もいて際立って高い比率を示している。[10] https://x.gd/55gtR |
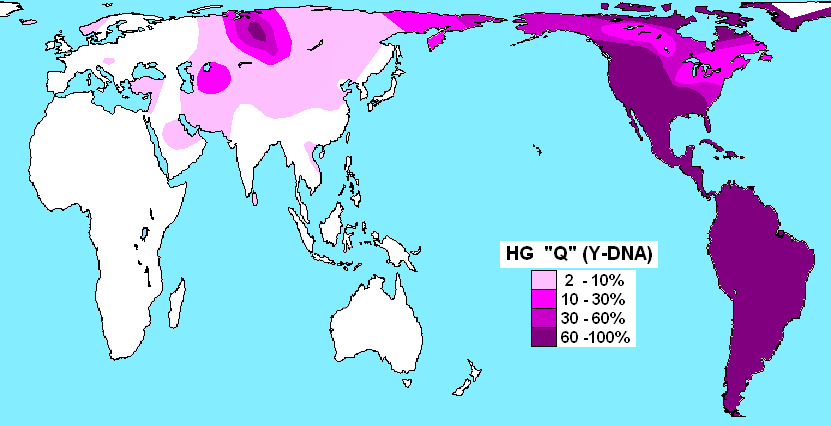 |
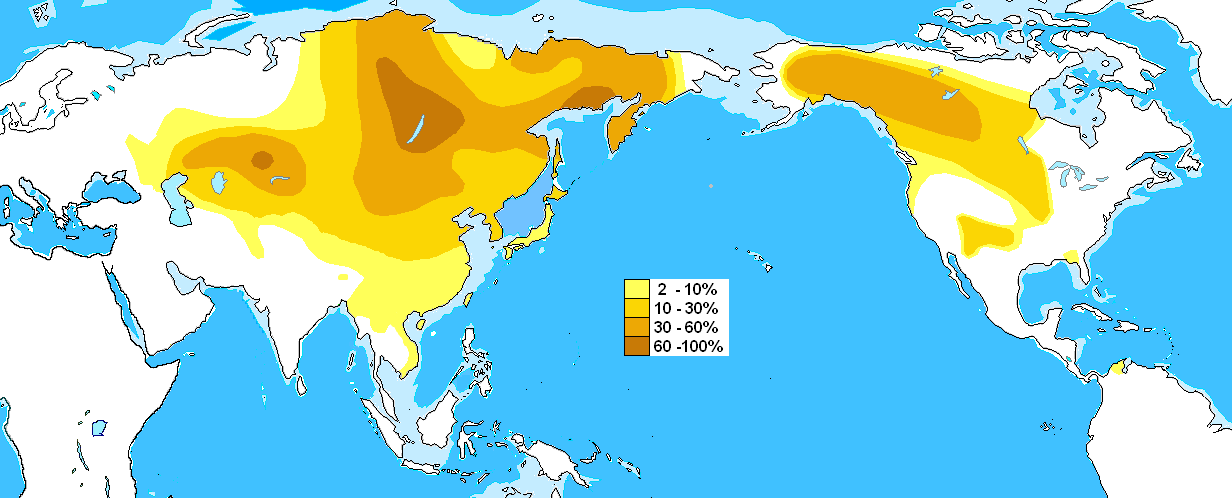
ハプログループQ (Y染色体)(ハプログループQ
(Yせんしょくたい)、英: Haplogroup Q
(Y-DNA))は、分子人類学において人類の父系を示すY染色体ハプログループ(型集団)の分類で、「M242, P36.2,
MEH2」のSNPの変異によって定義されるグループである。 概要 アメリカ先住民の Y 染色体ハプログループのほとんどはQ系統が占める。Q はイラン付近で17000-22000年前[1][2]または31,400年前[3]に発生したと考えられ、その後中央アジア、アルタイ山脈北辺を通り、 北シベリアのステップでマンモスなど大型哺乳類を狩りながら移動し、アメリカ大陸に移住していったと思われる。Q のホームランドに近いアフガニスタンのパシュトゥーン人では Q*が16%観察され[4]、中央シベリアのケット人でQ系統が90%以上を占める[5]など、道中に足跡を残してはいるが、ユーラシア大陸では総じて低 頻度である。しかしアメリカ大陸(先住民)において大いに繁栄している。アメリカ先住民にはその他 C2,R1 なども見られるが、Q系統が圧倒的である。 現代中国では全国男性人口の約2.8%がハプログループQに属し、そのほとんどが下位系統のQ-M120(全体の約2.48%[6])に分類されるが、Q -M346(約0.18%[7])、Q-L275(約0.09%[8])、Q-M25 > Q-L712(約0.04%[9])も稀に観察される。中国に於けるQ-M242の分布は著しく北方に偏っており、2011年に発表された論文では長江以 北の地域に居住する現代漢族男性の4.0%(34/853、30/853 = 3.52% Q-M120、3/853 = 0.35% Q-M346、1/853 = 0.12% Q-M25)がハプログループQに属す一方、長江以南の地域に居住する現代漢族男性のサンプルではハプログループQに属す者が全体の1.7% (15/876、14/876 = 1.60% Q-M120、1/876 = 0.11% Q-M346)に過ぎないという結果が得られた[10]。なお、ハプログループQは北方の少数民族である蒙古族(47/1521 = 3.09% Q、37/1521 = 2.43% Q-M120、8/1521 = 0.53% Q-M346 > Q-L330、2/1521 = 0.13% Q-M25 > Q-L712[11])や満洲族(76/2938 = 2.59% Q、67/2938 = 2.28% Q-M120 > Q-Z19154 > Q-F1626、5/2938 = 0.17% Q-M346、3/2938 = 0.10% Q-L275、1/2938 = 0.03% Q-M25 > Q-L712[12])でも観察されるが、むしろ華北の漢族男性の総人口に占めるハプログループQの割合が大きい。 現代韓国に於いてハプログループQは約1.7%(7/506 = 1.4%、1.6%、1/56 = 1.8%、18/1006 = 1.8%、1/45 = 2.2%、2/55 = 3.6%)の男性に観察される。2022年に発表された論文によると、群山市堂北里にある甕棺墓(年代は出土品の比較により六世紀中葉即ち百濟時代のもの と推定されている)に埋葬された人骨6体のうち、3体がハプログループQに属す男性だという結果が得られた。[13](残る3体のうち、2体が女性のもの で、1体がハプログループO1b2に属す男性のものだという。) 現代日本ではハプログループQは約0.40%(約250人に1人)という、ごく低頻度で観察されている。Hammer et al. (2006)による研究では、259人中1人(0.39%)がハプログループQ-P36に属しており、その1人は静岡県にて採取されたサンプルのもの。 [14] 野中氏等(2007年)による研究では、263人中1人(0.38%)がハプログループQ-M120に属しており、その1人は埼玉県にて採取されたサンプ ルのもの。[15] 宮崎県在住者1,677名及び熊本県在住者25名を対象とした研究では1,702人中7人(0.41%)がハプログループQ-P36.2に属すという結果 が得られた。[16] 徳島県(57人)+茨城県(50人)+山口県(44人)+大阪府或いは東京都(6人)にて採取された日本人157名を対象とした韓国の研究者による論文で は、ハプログループQ-M242に属す者は1人も観察されなかった。[17] 東京都にて採取されたJPT(Japanese in Tokyo, Japan)という1000人ゲノムプロジェクトのサンプルでは、56人中0人(0%)で、ハプログループQに属す者が一人もいないという結果が得られ た。[18] 日本人を対象としたその他の研究では出身地等詳しく発表されていないが、ハプログループQの比率は以下の通り: 567人中3人(0.53%)、[19] 432人中2人(0.46%)、[20] 47人中0人(0%)[21]。 https://x.gd/o4GPF |
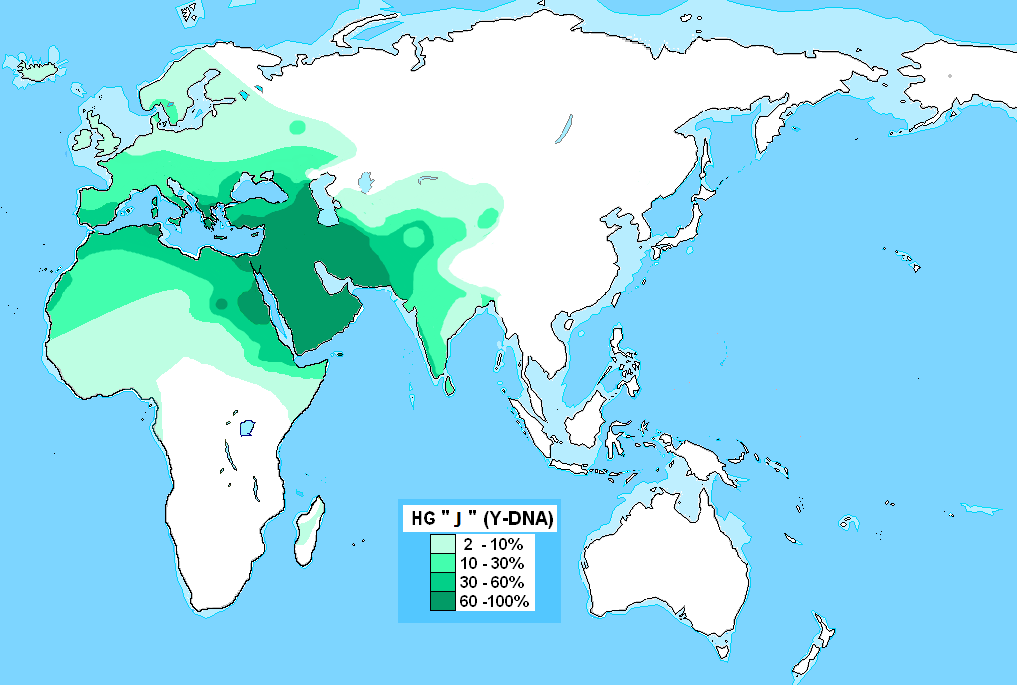 |
ハプログループJ-M304
(Y染色体)(ハプログループJ-M304〈Yせんしょくたい〉)、系統名称ハプログループJとは、分子人類学で用いられる、人類のY染色体ハプログルー
プの分類のうち、IJのサブクレード(細分岐)の一つで、「12f2.1, L134, M304, P209, S6/L60, S34,
S35」の変異で定義づけられる系統である。約48,000年前に誕生した。 下位系統 大きくJ1とJ2にわかれる。J1はアラブ人に多くみられ、アラビア半島や北アフリカで多い。J2は肥沃な三日月地帯からトルコ、ヨーロッパの地中海沿 岸、中央アジア、南アジアなどに多い。J1は南東人種、J2はアルメノイドの分布と概ね相関している。コーカサスでは民族や地域間の差が大きいが、全体的 に見ればJ1もJ2も多い地方の一つとして挙げられる。 J J* - J1 L255, L321/PF4646, M267/PF4782 J2 M172/Page28/PF4908, L228/PF4895/S321 (系統はHaplogroup Tree 2015 Version: 10.26 Date: 10 April 2015による。) https://x.gd/MLLiL |
●Y染色体ハプログループ移動の想定図
出典:崎谷満『DNA・考古・言語の学際研究が示す新・日本列島史 日本人集団・日本語の成立史』(勉誠出版 2009年)
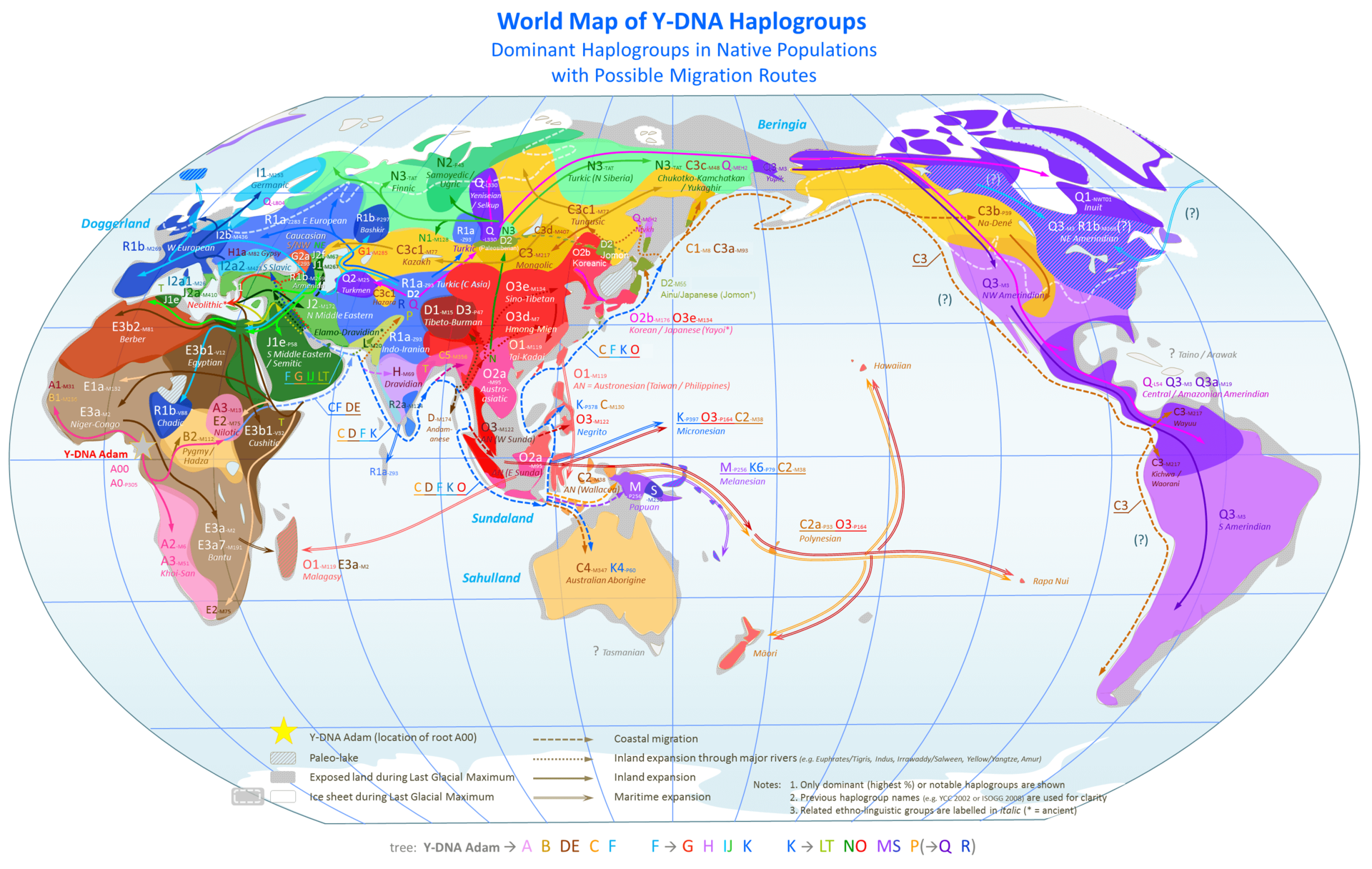
Y染色体ハプログループの世界拡散を表す想定地図(出典「Y染色体ハプログループ」)
用語解説
リンク
文献
その他の情報
Copyleft, CC, Mitzub'ixi Quq Chi'j, 1996-2099