
次世代シーケンシング
Next-Generation Sequencing (NGS), or
Massive parallel sequencing (MGS)

☆ 次世代シーケンシング(NGS)、大規模並列シーケンシング、ディープシーケンシングは、ゲノム研究に革命をもたらしたDNAシーケンシング技術を説明す る関連用語である。NGSを使用すれば、ヒトゲノム全体を1日以内に配列決定することができる。これに対し、ヒトゲノム解読に使用された従来のサンガー シークエンシング技術では、最終草案の完成までに10年以上を要した。ゲノム研究では、NGSが従来のサンガーシークエンシングに取って代わるケースがほ とんどであるが、臨床現場での日常的な使用にはまだ至っていない(→「ゲノミクス」)。
★以下は、まずは、この世代の直前の、第三世代シークエンシングについて解説する。
★【関連項目】「サンガー・シーケンシング(第一世代シーケンシング)」→「大規模並列シーケンシング(第二世代シーケンシング)」→「第三世代シーケンシング」→「次世代シーケンシング」
++
| DNA(デオキシリボ核酸)は、その構造内に
生命を創造し維持する生命システム全てに影響を及ぼす分子装置であるタンパク質やノンコーディングRNAの構築に必要なコードをもつ生命の青写真ともいえ
るものです。DNA配列を理解することで、研究者がRNAだけでなくタンパク質の構造や機能を解明することが可能となり、疾患の根底にある原因を理解する
ことが可能となってきました。次世代シーケンシング(NGS)は数千から数百万もの
DNA分子を同時に配列決定可能な強力な基盤技術です。次世代シーケンシングでは、複数個体を同時に配列決定できるなど高度かつ高速な処理が可能であるこ
とから、個の医療、遺伝性疾患、および臨床診断学といった分野に変革をもたらしています。 https://www.cosmobio.co.jp/support/technology/a/next-generation-sequencing-introduction-apb.asp |
 An Illumina HiSeq 2000 sequencing machine |
| 【旧世代のシークエンシンング】 サンガーシーケンシングでは高精度のDNA依存性ポリメラーゼを利用し、単一鎖DNA鋳型に対して相補的なコピーを産生します(1, 2, 3)。各反応において、鋳型に相補的なひとつのプライマーがその3'末端よりDNA合成反応を開始します。デオキシヌクレオチドまたは核酸はDNA単量体 ですが、これが次々に鋳型依存的に加えられ、伸展しているプライマー末端の3'ヒドロキシルと取り入れられてくる核酸の5'三リン酸塩基との間にリン酸ジ エステル結合が形成されます(図.1)(1)。 各反応にはそれぞれ各DNA塩基(つまり、A, G, T, およびC)に対応する4種のジデオキシヌクレオチドの混合物が含まれています。これらのジデオキシヌクレオチドはDNA単量体と類似しており伸展鎖に組込 まれますが、天然のデオキシヌクレオチドとは以下の2つの点が異なります。 さらなるDNA伸展に必要な3'ヒドロキシル基が欠損しており、DNA分子に組込まれると鎖の産生が停止する。 各ジデオキシヌクレオチドには独自の蛍光染色が付加されており、DNA配列を自動的に検出できる(3, 4, 5)。 反応ごとに多数の異なる鎖長のDNA断片コピーが作製され、この伸展は鋳型分子のヌクレオチド位の何れかにおいて対応するジデオキシヌクレオチドにより終 止されます(図.1)。反応混合液をシーケンシング機器にロードする際は、手動で平板ゲル上にロードするか毛細管を用いて自動でロードし、電気泳動を行っ てDNA分子を大きさで分類します。DNA配列はジデオキシヌクレオチドがゲルを通過する際にその蛍光発光を読み取ります(図.2)(5)。近年のサン ガーシーケンシング機器はキャピラリーを基盤とした自動化電気泳動を利用しており、通常、8~96シーケンシング反応を同時に分析することができます。 https://www.cosmobio.co.jp/support/technology/a/next-generation-sequencing-introduction-apb.asp |
★前史 「1975年、サンガーとアラン・コールソンは、DNAポリメラーゼと放射性標識ヌクレオチドを用いた塩基配列決定法を発表し、これをプラス・マイナス法 と呼んだ[21][22]。これらはポリアクリルアミドゲル上の電気泳動(ポリアクリルアミドゲル電気泳動と呼ばれる)によって分画され、オートラジオグ ラフィーを用いて可視化された。この方法では一度に80塩基までの塩基配列を決定することができ、大きな進歩であったが、それでも非常に手間のかかるもの であった。それでも1977年、彼のグループは一本鎖のバクテリオファージφX174の5,386ヌクレオチドのほとんどを配列決定することができ、初め てDNAベースのゲノムを完全に配列決定した。プラス・マイナス法を改良した結果、連鎖終結法、すなわちサンガー法(下記参照)が生まれ、その後の四半世 紀にわたる研究で最も広く使われたDNA配列決定、ゲノムマッピング、データ保存、バイオインフォマティクス解析の技術の基礎となった。[25]同年、 ハーバード大学のウォルター・ギルバートとアラン・マクサムは、既知の塩基におけるDNAの優先的切断を伴う、DNA配列決定のマクサム・ギルバート法 (化学的方法としても知られる)を独自に開発したが、これはあまり効率的な方法ではなかった[26][27]。核酸の配列決定における画期的な業績によ り、ギルバートとサンガーは、ポール・バーグ(組み換えDNA)と共に1980年のノーベル化学賞の半分を受賞した」(→「ゲノミクス」) |
| Massive parallel sequencing or
massively parallel sequencing is any of several high-throughput
approaches to DNA sequencing using the concept of massively parallel
processing; it is also called next-generation sequencing (NGS) or
second-generation sequencing. Some of these technologies emerged
between 1993 and 1998 [1][2][3][4][5] and have been commercially
available since 2005. These technologies use miniaturized and
parallelized platforms for sequencing of 1 million to 43 billion short
reads (50 to 400 bases each) per instrument run. Many NGS platforms differ in engineering configurations and sequencing chemistry. They share the technical paradigm of massive parallel sequencing via spatially separated, clonally amplified DNA templates or single DNA molecules in a flow cell. This design is very different from that of Sanger sequencing—also known as capillary sequencing or first-generation sequencing—which is based on electrophoretic separation of chain-termination products produced in individual sequencing reactions.[6] This methodology allows sequencing to be completed on a larger scale.[7] |
大規模並列シーケンシングまたは大規模並列シーケンシングは、大規模並
列処理の概念を用いたDNAシーケンシングのいくつかの高処理アプローチのうちの1つである。次世代シーケンシング(NGS)または第2世代シーケンシン
グとも呼ばれる。これらの技術の一部は1993年から1998年の間に登場し[1][2][3][4][5]、2005年以降は商業的に利用可能となって
いる。これらの技術では、小型化および並列化されたプラットフォームを使用し、1回の実行につき100万から430億の短いリード(各50から400塩
基)のシーケンスを行う。 多くのNGSプラットフォームは、エンジニアリング構成やシーケンス化学が異なる。それらは、フローセル内の空間的に分離されたクローン増幅DNAテンプ レートまたは単一DNA分子を介した大規模並列シーケンスという技術的パラダイムを共有している。この設計は、個々のシーケンス反応で生成された鎖停止産 物の電気泳動分離に基づくサンガーシーケンス(キャピラリーシーケンスまたは第一世代シーケンスとも呼ばれる)の設計とは大きく異なる。[6] この方法論により、より大規模なシーケンスが可能になる。[7] |
| Maxam–Gilbert
sequencing Third-generation sequencing |
|
| NGS platforms DNA sequencing with commercially available NGS platforms is generally conducted with the following steps. First, DNA sequencing libraries are generated by clonal amplification by PCR in vitro. Second, the DNA is sequenced by synthesis, such that the DNA sequence is determined by the addition of nucleotides to the complementary strand rather than through chain-termination chemistry. Third, the spatially segregated, amplified DNA templates are sequenced simultaneously in a massively parallel fashion without the requirement for a physical separation step. These steps are followed in most NGS platforms, but each utilizes a different strategy.[8] NGS parallelization of the sequencing reactions generates hundreds of megabases to gigabases of nucleotide sequence reads in a single instrument run. This has enabled a drastic increase in available sequence data and fundamentally changed genome sequencing approaches in the biomedical sciences.[9] Newly emerging NGS technologies and instruments have further contributed to a significant decrease in the cost of sequencing nearing the mark of $1000 per genome sequencing.[10][11] As of 2014, massively parallel sequencing platforms are commercially available and their features are summarized in the table. As the pace of NGS technologies is advancing rapidly, technical specifications and pricing are in flux. 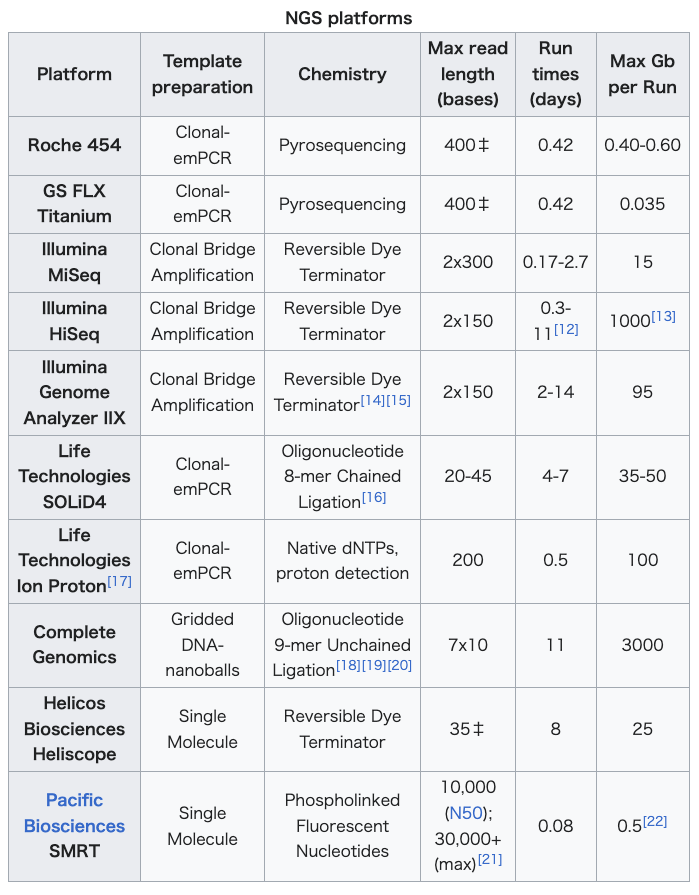 Run times and gigabase (Gb) output per run for single-end sequencing are noted. Run times and outputs approximately double when performing paired-end sequencing. ‡Average read lengths for the Roche 454 and Helicos Biosciences platforms.[23] |
NGSプラットフォーム 市販のNGSプラットフォームを用いたDNAシークエンシングは、一般的に以下の手順で行われる。まず、インビトロでのPCRによるクローン増幅により、 DNAシークエンシングライブラリが生成される。次に、DNAは合成によりシークエンシングされ、DNA配列は、鎖終結化学反応ではなく相補鎖へのヌクレ オチドの付加により決定される。第三に、空間的に分離された増幅されたDNAテンプレートは、物理的な分離ステップを必要とせずに、大規模な並列方式で同 時に配列決定される。これらのステップはほとんどのNGSプラットフォームで踏襲されているが、それぞれ異なる戦略が採用されている。 NGSのシーケンス反応の並列化により、1回の装置実行で数百メガベースからギガベースのヌクレオチド配列リードが生成される。これにより、利用可能な シーケンスデータの大幅な増加が可能になり、生物医学科学におけるゲノムシーケンスのアプローチが根本的に変化した。[9] 新たに登場したNGS技術と装置は、シーケンスコストの大幅な削減にさらに貢献し、ゲノムシーケンス1回あたり1000ドルという目標に近づいている。 [10][11] 2014年現在、超並列シーケンシングプラットフォームは市販されており、その特徴は表にまとめられている。NGS技術の進歩は急速であるため、技術仕様 や価格設定は流動的である。  シングルエンドシーケンスの実行時間と1回の実行ごとのギガベース(Gb)出力が記載されている。ペアエンドシーケンスを実行すると、実行時間と出力は約 2倍になる。‡ ロシュ454およびHelicosバイオサイエンスプラットフォームの平均リード長。[23] |
| Template preparation methods for NGS Two methods are used in preparing templates for NGS reactions: amplified templates originating from single DNA molecules, and single DNA molecule templates. For imaging systems which cannot detect single fluorescence events, amplification of DNA templates is required. The three most common amplification methods are emulsion PCR (emPCR), rolling circle and solid-phase amplification. The final distribution of templates can be spatially random or on a grid. Emulsion PCR In emulsion PCR methods, a DNA library is first generated through random fragmentation of genomic DNA. Single-stranded DNA fragments (templates) are attached to the surface of beads with adaptors or linkers, and one bead is attached to a single DNA fragment from the DNA library. The surface of the beads contains oligonucleotide probes with sequences that are complementary to the adaptors binding the DNA fragments. The beads are then compartmentalized into water-oil emulsion droplets. In the aqueous water-oil emulsion, each of the droplets capturing one bead is a PCR microreactor that produces amplified copies of the single DNA template.[24][25][26] Gridded rolling circle nanoballs Amplification of a population of single DNA molecules by rolling circle amplification in solution is followed by capture on a grid of spots sized to be smaller than the DNAs to be immobilized.[27][28][29][30] DNA colony generation (Bridge amplification) Forward and reverse primers are covalently attached at high-density to the slide in a flow cell. The ratio of the primers to the template on the support defines the surface density of the amplified clusters. The flow cell is exposed to reagents for polymerase-based extension, and priming occurs as the free/distal end of a ligated fragment "bridges" to a complementary oligo on the surface. Repeated denaturation and extension results in localized amplification of DNA fragments in millions of separate locations across the flow cell surface. Solid-phase amplification produces 100–200 million spatially separated template clusters, providing free ends to which a universal sequencing primer is then hybridized to initiate the sequencing reaction.[24][25] This technology was filed for a patent in 1997 from Glaxo-Welcome's Geneva Biomedical Research Institute (GBRI), by Pascal Mayer, Eric Kawashima, and Laurent Farinelli,[4][5] and was publicly presented for the first time in 1998.[31] In 1994 Chris Adams and Steve Kron filed a patent on a similar, but non-clonal, surface amplification method, named “bridge amplification”[3] adapted for clonal amplification in 1997 by Church and Mitra.[27][28] Single-molecule templates Protocols requiring DNA amplification are often cumbersome to implement and may introduce sequencing errors. The preparation of single-molecule templates is more straightforward and does not require PCR, which can introduce errors in the amplified templates. AT-rich and GC-rich target sequences often show amplification bias, which results in their underrepresentation in genome alignments and assemblies. Single molecule templates are usually immobilized on solid supports using one of at least three different approaches. In the first approach, spatially distributed individual primer molecules are covalently attached to the solid support. The template, which is prepared by randomly fragmenting the starting material into small sizes (for example,~200–250 bp) and adding common adapters to the fragment ends, is then hybridized to the immobilized primer. In the second approach, spatially distributed single-molecule templates are covalently attached to the solid support by priming and extending single-stranded, single-molecule templates from immobilized primers. A common primer is then hybridized to the template. In either approach, DNA polymerase can bind to the immobilized primed template configuration to initiate the NGS reaction. Both of the above approaches are used by Helicos BioSciences. In a third approach, spatially distributed single polymerase molecules are attached to the solid support, to which a primed template molecule is bound. This approach is used by Pacific Biosciences. Larger DNA molecules (up to tens of thousands of base pairs) can be used with this technique and, unlike the first two approaches, the third approach can be used with real-time methods, resulting in potentially longer read lengths. |
NGSのためのテンプレート準備方法 NGS反応用のテンプレートを準備するには、2つの方法が用いられる。1つは、単一DNA分子から増幅されたテンプレート、もう1つは、単一DNA分子テ ンプレートである。単一の蛍光事象を検出できないイメージングシステムでは、DNAテンプレートの増幅が必要である。最も一般的な増幅方法としては、エマ ルジョンPCR(emPCR)、ローリングサークル増幅、固相増幅の3つがある。テンプレートの最終的な配置は、空間的にランダムでもグリッド上でもよ い。 エマルジョン PCR エマルジョン PCR 法では、まずゲノム DNA をランダムに断片化して DNA ライブラリを生成する。一本鎖 DNA 断片(テンプレート)をアダプターまたはリンカーとともにビーズの表面に付着させ、DNA ライブラリから得た一本の DNA 断片に 1 つのビーズを付着させる。ビーズの表面には、DNA断片を結合するアダプターと相補的な配列を持つオリゴヌクレオチドプローブが結合している。その後、 ビーズは水相と油相のエマルション小滴に区画化される。水相と油相のエマルション中では、各小滴は1つのビーズを捕捉しており、これは1つのDNAテンプ レートを増幅したコピーを生成するPCRマイクロリアクターである。[24][25][26] グリッドローリングサークルナノボール 溶液中でのローリングサークル増幅による単一DNA分子の増幅は、固定化されるDNAよりも小さいサイズのスポットのグリッドへの捕捉が続く。[27][28][29][30] DNAコロニー生成(ブリッジ増幅 フォワードおよびリバースプライマーはフローセル内のスライドに高密度で共有結合する。支持体上のテンプレートに対するプライマーの比率は、増幅されたク ラスターの表面密度を決定する。フローセルは、ポリメラーゼをベースとする伸長反応用の試薬にさらされ、ライゲーションされた断片の遊離末端/遠位末端が 表面上の相補的オリゴに「橋渡し」されることでプライミングが行われる。変性と伸長を繰り返すことで、フローセル表面の数百万の別々の位置でDNA断片の 局所的な増幅が起こる。固相増幅により、1億から2億の空間的に分離したテンプレートクラスターが生成され、ユニバーサルシーケンスプライマーがハイブリ ダイズする遊離末端が提供される。これにより、シーケンス反応が開始される。[24][25] この技術は、1997年にグラクソ・ウェルカムのジュネーブ生物医学研究所(GBRI)のパスカル・メイヤー、エリック・カワシマ、ローラン・ファリネッ リにより特許出願された であり、1998年に初めて公に発表された。[31] 1994年、クリス・アダムスとスティーブ・クロンは、クローン増幅ではないが類似した表面増幅法である「ブリッジ増幅」[3]の特許を出願した。この方 法は、1997年にチャーチとミトラによってクローン増幅用に改良された。[27][28] 単一分子テンプレート DNA増幅を必要とするプロトコルは、実施するのが煩雑であることが多く、配列決定エラーを引き起こす可能性もある。単一分子テンプレートの準備はより簡 単であり、増幅テンプレートにエラーを引き起こす可能性のあるPCRを必要としない。ATに富む領域やGCに富む領域の標的配列は、増幅バイアスを示すこ とが多く、ゲノムのアラインメントやアセンブリにおいて過小評価される結果となる。通常、単一分子テンプレートは、少なくとも3つの異なるアプローチのう ちの1つを使用して、固体支持体に固定化される。最初の方法では、空間的に分散した個々のプライマー分子が固体支持体に共有結合で固定される。テンプレー トは、出発材料をランダムに断片化して小さなサイズ(例えば、200~250bp程度)にし、断片末端に共通アダプターを付加して調製され、その後、固定 化されたプライマーにハイブリダイズされる。2つ目のアプローチでは、空間的に分散した単一分子テンプレートが、固定化プライマーから単鎖の単一分子テン プレートをプライミングして伸長させることにより、固相支持体に共有結合で固定される。次に、共通プライマーがテンプレートにハイブリダイズする。いずれ のアプローチでも、DNAポリメラーゼは固定化されたプライミングされたテンプレート構造に結合し、NGS反応を開始することができる。上記の2つのアプ ローチは、Helicos BioSciences社によって使用されている。第3の手法では、空間的に分散した単一のポリメラーゼ分子が、プライミングされたテンプレート分子が結 合した固体支持体に結合される。この手法はパシフィック・バイオサイエンシズ社によって使用されている。この技術では、より大きなDNA分子(数万塩基対 まで)を使用することができ、最初の2つの手法とは異なり、第3の手法はリアルタイム法と併用できるため、より長いリード長を実現できる可能性がある。 |
| Sequencing approaches Sequencing by synthesis The objective for sequential sequencing by synthesis (SBS) is to determine the sequencing of a DNA sample by detecting the incorporation of a nucleotide by a DNA polymerase. An engineered polymerase is used to synthesize a copy of a single strand of DNA and the incorporation of each nucleotide is monitored. The principle of sequencing by synthesis was first described in 1993 [1] with improvements published some years later.[32] The key parts are highly similar for all embodiments of SBS and include (1) amplification of DNA to enhance the subsequent signal and to attach the DNA to be sequenced to a solid support, (2) generation of single stranded DNA on the solid support, (3) incorporation of nucleotides using an engineered polymerase and (4) detection of the incorporation of nucleotide. Then steps 3-4 are repeated and the sequence is assembled from the signals obtained in step 4. This principle of sequencing-by-synthesis has been used for almost all massive parallel sequencing instruments, including 454, PacBio, IonTorrent, Illumina and MGI. Pyrosequencing The principle of Pyrosequencing was first described in 1993 [1] by combining a solid support with an engineered DNA polymerase lacking 3´to 5´exonuclease activity (proof-reading) and luminescence real-time detection using the firefly luciferase. All the key concepts of sequencing by synthesis were introduced, including (1) amplification of DNA to enhance the subsequent signal and attach the DNA to be sequenced (template) to a solid support, (2) generation of single stranded DNA on the solid support (3) incorporation of nucleotides using an engineered polymerase and (4) detection of the incorporated nucleotide by light detection in real-time. In a follow-up article,[2] the concept was further developed and in 1998, an article [32] was published in which the authors showed that non-incorporated nucleotides could be removed with a fourth enzyme (apyrase) allowing sequencing by synthesis to be performed without the need for washing away non-incorporated nucleotides. Sequencing by reversible terminator chemistry This approach uses reversible terminator-bound dNTPs in a cyclic method that comprises nucleotide incorporation, fluorescence imaging and cleavage. A fluorescently-labeled terminator is imaged as each dNTP is added and then cleaved to allow incorporation of the next base. These nucleotides are chemically blocked such that each incorporation is a unique event. An imaging step follows each base incorporation step, then the blocked group is chemically removed to prepare each strand for the next incorporation by DNA polymerase. This series of steps continues for a specific number of cycles, as determined by user-defined instrument settings. The 3' blocking groups were originally conceived as either enzymatic[33] or chemical reversal[14][15] The chemical method has been the basis for the Solexa and Illumina machines. Sequencing by reversible terminator chemistry can be a four-colour cycle such as used by Illumina/Solexa, or a one-colour cycle such as used by Helicos BioSciences. Helicos BioSciences used “virtual Terminators”, which are unblocked terminators with a second nucleoside analogue that acts as an inhibitor. These terminators have the appropriate modifications for terminating or inhibiting groups so that DNA synthesis is terminated after a single base addition.[25][34][35] Sequencing-by-ligation mediated by ligase enzymes In this approach, the sequence extension reaction is not carried out by polymerases but rather by DNA ligase and either one-base-encoded probes or two-base-encoded probes. In its simplest form, a fluorescently labelled probe hybridizes to its complementary sequence adjacent to the primed template. DNA ligase is then added to join the dye-labelled probe to the primer. Non-ligated probes are washed away, followed by fluorescence imaging to determine the identity of the ligated probe. The cycle can be repeated either by using cleavable probes to remove the fluorescent dye and regenerate a 5′-PO4 group for subsequent ligation cycles (chained ligation[16][36]) or by removing and hybridizing a new primer to the template (unchained ligation[18][19]). Phospholinked Fluorescent Nucleotides or Real-time sequencing Pacific Biosciences is currently leading this method. The method of real-time sequencing involves imaging the continuous incorporation of dye-labelled nucleotides during DNA synthesis: single DNA polymerase molecules are attached to the bottom surface of individual zero-mode waveguide detectors (Zmw detectors) that can obtain sequence information while phospholinked nucleotides are being incorporated into the growing primer strand. Pacific Biosciences uses a unique DNA polymerase which better incorporates phospholinked nucleotides and enables the resequencing of closed circular templates. While single-read accuracy is 87%, consensus accuracy has been demonstrated at 99.999% with multi-kilobase read lengths.[37][38] In 2015, Pacific Biosciences released a new sequencing instrument called the Sequel System, which increases capacity approximately 6.5-fold.[39][40] |
シーケンスアプローチ 合成によるシーケンス 合成シーケンス(SBS)の目的は、DNAポリメラーゼによるヌクレオチドの取り込みを検出することで、DNAサンプルのシーケンスを決定することであ る。 設計されたポリメラーゼが、DNAの単鎖のコピーを合成するために使用され、各ヌクレオチドの取り込みがモニターされる。合成シーケンシングの原理は、 1993年に初めて発表され[1]、その後数年間に改良が加えられた。[32] SBSのすべての実施態様において、重要な部分は非常に類似しており、(1) その後のシグナルを増幅し、 シグナルを増幅し、配列決定するDNAを固体支持体に結合させる、(2) 固体支持体上で一本鎖DNAを生成する、(3) 改変ポリメラーゼを使用してヌクレオチドを取り込む、(4) ヌクレオチドの取り込みを検出する。その後、ステップ3から4が繰り返され、ステップ4で得られたシグナルから配列が組み立てられる。このシーケンス・バ イ・シンセシスの原理は、454、PacBio、IonTorrent、Illumina、MGIなど、ほぼすべての大量並列シーケンシング装置で使用さ れている。 パイロシークエンシング パイロシークエンシングの原理は、1993年に初めて発表された[1]。3´から5´エキソヌクレアーゼ活性(校正機能)を欠くように設計されたDNAポ リメラーゼと、ホタルルシフェラーゼを用いた発光リアルタイム検出を組み合わせたものである。合成によるシーケンシングのすべての主要な概念が紹介され た。これには、(1) その後のシグナルを増幅し、シーケンスするDNA(テンプレート)を固相に結合させるためのDNAの増幅、(2) 固相上での一本鎖DNAの生成、(3) 改良型ポリメラーゼによるヌクレオチドの取り込み、(4) 取り込まれたヌクレオチドのリアルタイムでの光検出による検出、が含まれる。その後の記事[2]では、このコンセプトがさらに発展し、1998年には、第 4の酵素(アピラーゼ)によって非取り込みヌクレオチドを除去できることが示された論文[32]が発表された。これにより、非取り込みヌクレオチドを洗い 流す必要なく合成によるシーケンスを行うことが可能になった。 可逆的ターミネーター化学によるシーケンス このアプローチでは、可逆的ターミネーターに結合したdNTPsを、ヌクレオチド取り込み、蛍光イメージング、切断からなる循環的な方法で使用する。各 dNTPが添加され、次に次の塩基の取り込みを可能にするために切断されると、蛍光標識されたターミネーターがイメージ化される。これらのヌクレオチド は、各取り込みが唯一のイベントとなるように化学的にブロックされる。各塩基取り込みステップの後にイメージングステップが続き、その後、ブロックされた グループが化学的に除去され、DNAポリメラーゼによる次の取り込みに備えて各鎖が準備される。この一連のステップは、ユーザー定義の機器設定によって決 定される特定のサイクル数だけ継続される。3'ブロック基は、当初は酵素[33]または化学的[14][15]のいずれかで解除する方法として考案され た。化学的方法は、SolexaやIlluminaの機械の基礎となっている。可逆的ターミネーター化学による配列決定は、 Illumina/Solexaで使用されているような4色のサイクル、またはHelicos BioSciencesで使用されているような1色のサイクルが可能である。Helicos BioSciencesは「仮想ターミネーター」を使用していた。これは阻害剤として働く2番目のヌクレオシドアナログを持つ、ブロックされていないター ミネーターである。これらのターミネーターは、DNA合成を1塩基の付加後に停止させるために、停止または阻害基に適切な修飾が施されている。[25] [34][35] リガーゼ酵素によるライゲーション媒介シーケンシング このアプローチでは、配列の伸長反応はポリメラーゼではなく、DNAリガーゼと1塩基プローブまたは2塩基プローブによって行われる。最も単純な形では、 蛍光標識プローブがプライミングされたテンプレートの隣接する相補的配列にハイブリダイズする。次にDNAリガーゼが添加され、色素標識プローブをプライ マーに結合させる。ライゲーションされていないプローブは洗浄により除去され、続いて蛍光イメージングによりライゲーションされたプローブの同一性を決定 する。このサイクルは、切断可能なプローブを使用して蛍光色素を除去し、次のライゲーションサイクルのための5′-リン酸基を再生させる(連鎖ライゲー ション[16][36])か、または新しいプライマーをテンプレートから除去し、ハイブリダイズさせる(非連鎖ライゲーション[18][19])ことによ り、繰り返すことができる。 リン酸結合蛍光ヌクレオチドまたはリアルタイムシークエンシング パシフィック・バイオサイエンシズ社は現在、この方法で業界をリードしている。リアルタイムシークエンシングの手法では、DNA合成中に色素標識ヌクレオ チドを連続的に取り込む様子を画像化する。個々のDNAポリメラーゼ分子は、ゼロモード波導検出器(Zmw検出器)の底面に付着しており、伸長するプライ マー鎖にリン酸結合ヌクレオチドが取り込まれる際に、配列情報を取得することができる。パシフィック・バイオサイエンシズ社は、リン酸結合ヌクレオチドを よりよく取り込む独自のDNAポリメラーゼを使用しており、閉環状テンプレートの再シークエンシングを可能にしている。シングルリードの精度は87%であ るが、マルチキロベースリード長ではコンセンサス精度が99.999%であることが実証されている。[37][38] 2015年、パシフィック・バイオサイエンシズは、シーケンス能力を約6.5倍に高めた新シーケンス装置「Sequelシステム」を発表した。[39] [40] |
| Clinical metagenomic sequencing First-generation sequencing (Sanger sequencing) Third-generation sequencing RNA Velocity |
臨床メタゲノムシーケンス 第一世代シーケンサー(Sanger sequencing) 第三世代シーケンサー RNAの速度 |
| Illumina, Inc.
is an American biotechnology company, headquartered in San Diego,
California. Incorporated on April 1, 1998, Illumina develops,
manufactures, and markets integrated systems for the analysis of
genetic variation and biological function. The company provides a line
of products and services that serves the sequencing, genotyping and
gene expression, and proteomics markets, and serves more than 155
countries.[2] Illumina's customers include genomic research centers, pharmaceutical companies, academic institutions, clinical research organizations, and biotechnology companies.[1] History  Czarnik, Stuelpnagel, and Chee at their Illumina office in the summer of 1998 Illumina was founded in April 1998 by David Walt, Larry Bock, John Stuelpnagel, Anthony Czarnik, and Mark Chee. While working with CW Group, a venture-capital firm, Bock and Stuelpnagel uncovered what would become Illumina's BeadArray technology[3] at Tufts University and negotiated an exclusive license to that technology. In 1999, Illumina acquired Spyder Instruments (founded by Michal Lebl, Richard Houghten, and Jutta Eichler) for their technology of high-throughput synthesis of oligonucleotides. Illumina completed its initial public offering in July 2000.[4] Illumina began offering single nucleotide polymorphism (SNP) genotyping services in 2001 and launched its first system, the Illumina BeadLab, in 2002, using GoldenGate Genotyping technology. Illumina currently offers microarray-based products and services for an expanding range of genetic analysis sequencing, including SNP genotyping, gene expression, and protein analysis. Illumina's technologies are used by a broad range of academic, government, pharmaceutical, biotechnology, and other leading institutions around the globe. On January 26, 2007, the company completed the acquisition of the British company Solexa, Inc. for ~$650M.[5] Solexa was founded in June 1998 by Shankar Balasubramanian and David Klenerman to develop and commercialize genome-sequencing technology invented by the founders at the University of Cambridge. Solexa, Inc. was formed in 2005 when Solexa Ltd reversed into Lynx Therapeutics of Hayward.[6] Illumina also uses the DNA colony sequencing technology, invented in 1997 by Pascal Mayer and Laurent Farinelli [7] and which was acquired by Solexa in 2004 from Manteia Predictive Medicine. It is being used to perform a range of analyses, including whole genome resequencing, gene-expression analysis, and small ribonucleic acid (sRNA) analysis. In June 2009, Illumina announced the launch of their own Personal Full Genome Sequencing Service at a depth of 30X.[8] Until 2010, Illumina sold only instruments that were labeled "for research use only"; in early 2010, Illumina obtained FDA approval for its BeadXpress system to be used in clinical tests.[9][10] This was part of the company's strategy at the time to open its own CLIA lab and begin offering clinical genetic testing itself.[11] Illumina acquired Epicentre Biotechnologies, based in Madison, Wisconsin, on January 11, 2011.[12] On January 25, 2012, Hoffmann-La Roche made an unsolicited bid to buy Illumina for $44.50 per share or about $5.7 billion.[13][14] Roche tried other tactics, including raising its offer (to $51.00, for about $6.8 billion).[15] Illumina rejected the offer,[16] and Roche abandoned the offer in April.[17] In 2014, the company announced a multimillion-dollar product, HiSeq X Ten.[18][19] In January 2014, Illumina already held 70% of the market for genome-sequencing machines.[20] Illumina machines accounted for more than 90% of all DNA data produced.[21] In 2020, the company invested in the acquisition of the pre-commercial firm Enancio, which had developed a DNA data compression algorithm specifically targeting Illumina data capable of reducing storage footprint by 80% (e.g. 50 GB compressed to 10 GB).[22] On July 5, 2016, Jay Flatley, who had been CEO since 1999, assumed the role of executive chairman of the board of directors. Francis deSouza, who had been president of the company since 2013, took on the additional role of CEO.[23] In late 2015, Illumina spun off the company Grail, focused on blood testing for cancer tumors in the bloodstream. In 2017 Grail had planned to raise $1 billion in its second round of financing, and received funding from Bill Gates and Jeff Bezos investing $100 million in series A funding, and with Illumina maintaining a 20% holding share in Grail.[24] Grail is working with a blood test trial with over 120,000 women during scheduled mammogram visits in the states of Minnesota and Wisconsin, as well as a partnership with the Mayo Clinic. Grail uses Illumina sequencing technology for tests.[25] Grail planned to roll out the tests by 2019.[26] In September 2020, Illumina announced a proposed cash and stock deal to acquire Grail for $8 billion.[27][28] In November 2018, Illumina proposed the acquisition of Pacific Biosciences for $8.00 per share or around $1.2 billion in total.[29][30] In December 2019, the Federal Trade Commission (FTC) sued to block the acquisition.[31] The proposed deal was abandoned on January 2, 2020, with Illumina paying Pacific a $98 million termination fee.[32] In March 2021, the FTC sued to block Illumina's $7.1 billion vertical merger with Grail.[33][34] In July 2021, the European Commission opened an in-depth investigation into the Grail acquisition by Illumina.[35] Against the orders of active investigations by both the US FTC and the EU European Commission, Illumina publicly announced it had completed its acquisition of Grail on August 18, 2021.[36] The FTC urged Illumina to "unwind" the merger shortly after,[37] and in October 2021, the European Commission ordered Illumina to keep Grail a separate company[38] and adopted interim measures to prevent harm to competition, or face penalty payments up to 5% of their average daily turnover and/or fines up to 10% of their annual worldwide turnover under Articles 15 and 14 of the EU Merger Regulation respectively.[35] In September 2022, a US administrative judge ruled against the FTC's efforts to prevent the acquisition on antitrust grounds.[39] In April 2023, the FTC ordered Grail to be divested by Illumina.[40] In July 2023, the European Commission imposed a €432 million ($476 million) penalty on Illumina for closing the Grail acquisition without EU approval.[41] In September 2022, Illumina launched NovaSeq X and NovaSeq X Plus.[42] The NovaSeq X Plus can sequence 20,000 genomes per year, compared to 7,500 per year of Illumina's previous machines and generate up to 16 Tb of data per run.[42] The series includes redeveloped reagents, dyes, and polymerases which can be shipped at ambient temperature.[43] In June 2023, deSouza resigned as CEO of Illumina, and was replaced by interim CEO Charles Dadswell, the company's general counsel.[44] Also in June 2023, Hologic CEO Stephen Macmillan was named non-executive Chairman of the Board of Directors.[45] In September 2023, Agilent Technologies' senior vice president Jacob Thaysen was appointed CEO.[46] In October 2023, the European Commission ordered Grail to be divested from Illumina within the next twelve months.[47] Illumina said it would explore a third-party sale or a capital markets transaction if it fails to win its ongoing challenge in court.[48] |
イ
ルミナ(Illumina,
Inc.)は、アメリカ合衆国のカリフォルニア州サンディエゴに本社を置くバイオテクノロジー企業である。1998年4月1日に法人化されたイルミナは、
遺伝的多様性と生物学的機能の解析のための統合システムの開発、製造、販売を行っている。同社は、シーケンス、遺伝子タイピング、遺伝子発現、プロテオミ
クス市場向けの製品とサービスを提供しており、155カ国以上で事業を展開している。 イルミナの顧客には、ゲノム研究センター、製薬会社、学術機関、臨床研究機関、バイオテクノロジー企業などが含まれる。[1] 沿革  1998年夏、イルミナのオフィスにて、Czarnik、Stuelpnagel、Chee イルミナは、1998年4月にDavid Walt、Larry Bock、John Stuelpnagel、Anthony Czarnik、Mark Cheeによって設立された。ボックとステュエルプネゲルは、ベンチャーキャピタル企業CWグループに勤務していた際に、タフツ大学で後にイルミナの BeadArray技術となるものを発見し[3]、その技術の独占ライセンスの交渉を行った。1999年、イルミナはオリゴヌクレオチドの高処理合成技術 を獲得するために、スパイダー・インスツルメンツ社(ミハエル・レブル、リチャード・ホーテン、ユッタ・アイヒラーにより設立)を買収した。イルミナは 2000年7月に新規株式公開を完了した。[4] イルミナは2001年に一塩基多型(SNP)ジェノタイピングサービスの提供を開始し、2002年にはGoldenGateジェノタイピング技術を使用し た最初のシステムであるイルミナBeadLabを発売した。現在イルミナは、SNPジェノタイピング、遺伝子発現、タンパク質解析など、拡大する遺伝子解 析シーケンスの分野を対象に、マイクロアレイベースの製品およびサービスを提供している。イルミナの技術は、世界中の幅広い学術機関、政府機関、製薬会 社、バイオテクノロジー企業、その他の主要機関で使用されている。 2007年1月26日、同社はイギリスの企業であるソレクサ社を約6億5000万ドルで買収した。ソレクサ社は、1998年6月にシャンカル・バラスブラ マニアムとデビッド・クレナーマンによって、ケンブリッジ大学で発明されたゲノム配列決定技術の開発と商業化を目的として設立された。2005年、 Solexa Ltd.はヘイワードのLynx Therapeuticsに吸収合併され、Solexa, Inc.が設立された。[6]イルミナも、1997年にパスカル・メイヤーとローラン・ファレネリによって発明されたDNAコロニーシークエンシング技術 を使用している。[7]この技術は、2004年にManteia Predictive Medicineからソレクサが取得したものである。この技術は、全ゲノムの再シークエンシング、遺伝子発現解析、および小リボ核酸(sRNA)解析を含 む、さまざまな解析に使用されている。 2009年6月、イルミナは、深度30Xの独自のパーソナル全ゲノムシークエンシングサービスの開始を発表した。[8] 2010年までは、イルミナは「研究用のみ」と表示された機器のみを販売していたが、2010年初頭に、イルミナは臨床検査で使用する BeadXpressシステムについてFDAの承認を取得した。[9][10]これは、当時、同社がCLIAラボを独自に開設し、臨床遺伝子検査を自ら提 供し始めるという戦略の一環であった。[11] イルミナは2011年1月11日、ウィスコンシン州マディソンに拠点を置くEpicentre Biotechnologiesを買収した。[12] 2012年1月25日、ホフマン・ラ・ロシュはイルミナの株式1株あたり44.50ドル、総額約57億ドルでの買収を提案した。約57億ドルであった。 [13][14] ロシュは、買収額を51.00ドル(約68億ドル)に引き上げるなど、他の戦術を試みた。[15] イルミナは買収提案を拒否し、[16] ロシュは4月に買収提案を断念した。[17] 2014年、同社は数百万ドルの製品HiSeq X Tenを発表した。[18][19] 2014年1月時点で、イルミナはすでにゲノムシーケンシングマシンの市場の70%を占めていた。[20] イルミナの機械は、生成されたDNAデータの90%以上を占めていた 。2020年には、同社は非上場企業であるEnancioの買収に投資した。Enancioは、イルミナのデータを特にターゲットとしたDNAデータ圧縮 アルゴリズムを開発しており、ストレージのフットプリントを80%削減できる(例えば、50GBを10GBに圧縮できる)可能性がある。 2016年7月5日、1999年よりCEOを務めていたジェイ・フラットリーが取締役会の執行会長に就任した。2013年より社長を務めていたフランシス・デスーザがCEOを兼任することになった。 2015年後半、イルミナは血流中の癌腫瘍の血液検査に焦点を当てた企 業Grailをスピンオフさせた。2017年、Grailは2回目の資金調達ラウンドで10億ドルの調達を計画し、ビル・ゲイツとジェフ・ベゾスがシリー ズAの資金調達に1億ドルを投資し、イルミナはGrailの20%の株式を維持した Grailの株式を20%保有している。[24] Grailは、ミネソタ州とウィスコンシン州でマンモグラフィー検診を受ける予定の12万人以上の女性を対象とした血液検査試験を実施しているほか、メイ ヨー・クリニックとも提携している。Grailは検査にイルミナのシーケンシング技術を使用している。[25] Grailは2019年までに検査を展開する計画であった。[26] 2020年9月、イルミナはGrailを80億ドルで買収する現金および株式取引案を発表した。[27][28] 2018年11月、イルミナはパシフィック・バイオサイエンシズの買収を1株あたり8.00ドル、総額約12億ドルで提案した。[29][30] 2019年12月、連邦取引委員会(FTC)は買収を阻止するために提訴した。[31] 2020年1月2日、この提案された取引は中止され、イルミナはパシフィックに9800万ドルの解約料を支払った。[32] 2021年3月、FTCはイルミナによるGrailとの71億ドルの垂直統合を阻止するために提訴した。[33][34] 2021年7月、欧州委員会はイルミナによるGrail買収に関する詳細な調査を開始した。[35] 米国のFTCとEU欧州委員会の両方による積極的な調査の命令に反して イルミナは2021年8月18日にグレイルの買収を完了したことを公表した。[36] FTC は直後にイルミナに合併の「解消」を促し[37]、2021年10月には欧州委員会がイルミナにグレイルを別会社として維持するよう命じ[38]、 競争への悪影響を防ぐための暫定措置を採択し、EU合併規則第15条および第14条に基づき、それぞれ平均日次売上高の5%までの制裁金、および/または 年間世界売上高の10%までの罰金を科す可能性があるとした。[35] 2022年9月、米国の行政裁判官は、 独占禁止法上の理由から買収を阻止しようとしたFTCの取り組みを却下した。[39] 2023年4月、FTCはGrailをIlluminaから売却するよう命じた。[40] 2023年7月、欧州委員会はEUの承認なしにGrailの買収を完了したとして、Illuminaに4億3200万ユーロ(4億7600万ドル)の制裁 金を科した。[41] 2022年9月、イルミナはNovaSeq XとNovaSeq X Plusを発表した(→NextSeq 1000 & NextSeq 2000)。 [42] NovaSeq X Plusは、イルミナの以前の機種では年間7,500だったのに対し、年間20,000のゲノム配列決定が可能であり、1回の実行につき最大16テラバイ トのデータを生成できる。[42] このシリーズには、常温で出荷できる再開発された試薬、色素、およびポリメラーゼが含まれている。[43] 2023年6月、デスーザはイルミナのCEOを辞任し、同社の法務顧問であるチャールズ・ダッズウェルが暫定CEOに就任した。[44] 同じく2023年6月、ホロジックのCEOスティーブン・マクミランが取締役会の非常勤会長に就任した。[45] 2023年9月、アジレント・テクノロジーの上級副社長ジェイコブ・セイセンがCEOに任命された。[46] 2023年10月、欧州委員会は、今後12か月以内にイルミナからグレイルを売却するよう命じた。[47] イルミナは、現在係争中の裁判で敗訴した場合、第三者への売却または資本市場取引を検討するとしている。[48] |
| Acquisition history |
買収史(省略) |
| Products DNA sequencing Main articles: Illumina (Solexa) sequencing and Illumina dye sequencing Illumina MiSeq sequencer Illumina sells a number of high-throughput DNA sequencing systems, also known as DNA sequencers, based on technology developed by Solexa. The technology features bridge amplification to generate clusters and reversible terminators for sequence determination.[49][50] The technology behind these sequencing systems involves ligation of fragmented DNA to a chip, followed by primer addition and sequential fluorescent dNTP incorporation and detection. Depending on the kit used, according to the company the MiSeq Series generates up to 25 million reads per run.[51] With dual flow cells, the NextSeq 2000 generates up to 2.4 billion single reads per run[52] and the NovaSeq X Series generates up to 52 billion single reads per run.[53] Illumina uses next-generation sequencing, which is far faster and more efficient than traditional Sanger sequencing.[54] Illumina sequencers perform short-read sequencing, and are image based, utilizing Illumina dye sequencing.[54] This technology has a higher accuracy than long-read sequencing.[54] Flow cells MiSeq Flow Cell (Top) NovaSeq Flow Cell Illumina sequencing happens within the flow cells. These flow cells are small in size and are housed in the flow cell compartment. Flow cell clustering happens when a denatured DNA sample is placed in a flow cell. Primers already in the flow cell channel capture and bind to the ends of the short denatured DNA sample. Then, DNA polymerase is added and the DNA building blocks are introduced. This results in a newly synthesized strand constrained to the bottom of the flow cell. Next, the original template strand is washed out binding the newly synthesized strand to the other DNA sequence present on the surface. DNA polymerase and building blocks are introduced again forming a new strand. These steps are repeated until about 1,000 copies are made in a cluster.[54] |
製品 DNAシークエンシング 主な記事:イルミナ(ソレクサ)シークエンシングおよびイルミナ色素シークエンシング イルミナMiSeqシーケンサー イルミナは、ソレクサが開発した技術を基に、DNAシーケンサーとも呼ばれる多数のハイスループットDNAシーケンシングシステムを販売している。この技 術は、クラスター生成のためのブリッジ増幅と、配列決定のための可逆的ターミネーターを特徴とする。[49][50] これらのシーケンシングシステムを支える技術は、断片化されたDNAをチップに結合させ、続いてプライマーを添加し、蛍光dNTPを逐次的に取り込み、検 出するというものである。 使用するキットによって異なるが、同社によるとMiSeqシリーズは1回の実行につき最大2500万リードを生成する。[51]デュアルフローセルを使用 するNextSeq 2000は1回の実行につき最大24億のシングルリードを生成し[52]、NovaSeq Xシリーズは1回の実行につき最大520億のシングルリードを生成する リードを生成する。[53]イルミナは次世代シーケンシングを使用しており、これは従来のサンガーシーケンシングよりもはるかに高速で効率的である。 [54]イルミナシーケンサーはショートリードシーケンシングを行い、イメージベースで、イルミナ色素シーケンシングを利用している。[54]このテクノ ロジーは、ロングリードシーケンシングよりも精度が高い。[54] フローセル MiSeqフローセル(上) NovaSeqフローセル イルミナシーケンシングはフローセル内で実行される。これらのフローセルは小型で、フローセルコンパートメントに収容されている。変性DNAサンプルがフ ローセルに配置されると、フローセルのクラスタリングが開始される。フローセルチャネルにすでに存在するプライマーが、短い変性DNAサンプルの両端を捕 捉し結合する。次に、DNAポリメラーゼが添加され、DNAビルディングブロックが導入される。その結果、新たに合成された鎖がフローセルの底面に固定さ れる。次に、元のテンプレート鎖が洗い流され、新たに合成された鎖が表面に存在する他のDNA配列に結合する。DNAポリメラーゼとビルディングブロック が再び導入され、新たな鎖が形成される。これらのステップは、約1,000コピーがクラスター内で作成されるまで繰り返される。[54] |
| Litigation Czarnik suit against Illumina In 2005, co-founder and former Chief Scientific Officer Anthony Czarnik sued Illumina. In the case, Czarnik v. Illumina Inc., the trial court granted Illumina's motion to dismiss in part but allowed Czarnik's correction of inventorship claims to continue.[55] Cornell University and Life Technologies suit against Illumina In 2010, Cornell University and Life Technologies filed a lawsuit against Illumina, alleging that its microarray products infringed on eight patents held by the university and exclusively licensed to the start-up. The case was settled in April 2017 without any finding of fault. In September 2017 both parties asked to have the settlement reviewed, with Cornell accusing both Illumina and Life Technologies of misrepresentation and fraud.[56] Cornell claimed that ThermoFisher had promised to settle the suit with Illumina and asked for the Markman wording to be dropped so that it could file a subsequent suit involving other patents invented at Cornell. Instead of filing the suit, ThermoFisher and Illumina settled another lawsuit in California and secretly sublicensed those very same patents. In 2018, Dr. Monib Zirvi filed a lawsuit in the Southern District of New York against Illumina and some of its key employees claiming that they knowingly incorporated ideas and ZipCode DNA sequences invented in the Barany Lab in Illumina's patent applications. Although this suit was dismissed, it was only after Illumina and its attorneys claimed that some of those IP misappropriation were “storm warnings” and thus statutes of limitations had run out on those particular claims. Dr. Monib Zirvi also filed a FOIA case in New Jersey in 2020 for unredacted copies for key NIH grants that Illumina filed early in its existence. William Noon, an in-house attorney at Illumina, had filed a FOIA request for 4 of these key grants as well in January 2015. Patent infringement suits Illumina was a party in a patent lawsuit against competitor Ariosa Diagnostics. The litigation began in 2012 with Verinata Health filing suit against Ariosa. Illumina joined the suit after acquiring Verinata in 2013. Ariosa subsequently brought a counterclaim against Illumina.[57] The trial court granted summary judgment in favor of Ariosa, but the United States Court of Appeals for the Federal Circuit reversed.[58] Ariosa initially pursued an appeal to the Supreme Court of the United States, but the two parties resolved the dispute before the Court decided whether to take the case.[59] In February 2016, Illumina filed a lawsuit against Oxford Nanopore Technologies. Illumina claimed that Oxford Nanopore infringed its patents on the use of a biological nanopore, Mycobacterium smegmatis porinA (MspA), for sequencing systems.[60][61] In August 2016 the parties settled their lawsuit.[62] In February 2020, Illumina filed a patent infringement suit against BGI relating to its "CoolMPS" sequencing products.[63] In return BGI has filed patent infringement lawsuits for violation of federal antitrust and California unfair competition laws, claiming use of "fraudulent behavior" to obtain or enforce sequencing patents that it has asserted against BGI, preventing the firm from entering the US market.[64] However, in May 2022, Illumina was ordered to pay $333 million to a U.S. unit of BGI in California for infringing two patents of DNA-sequencing systems. The jury of the case also said that Illumina willfully infringed the patents, and that their former accusation of BGI's infringement was invalid.[65] On May 6, 2022, a jury in the U.S. District Court for the District of Delaware rendered a verdict that Illumina willfully infringed two patents owned by Complete Genomics, and awarded approximately $334 million to CGI in past damages. The jury also invalidated three patents owned by Illumina.[66] Trade secrets suit against Eltoukhy and Talasaz In March 2022, Illumina sued Helmy Eltoukhy and Amir Talasaz, the co-founders of Guardant, over stealing trade secrets.[67] Guardant called the lawsuit "frivolous and retaliatory" and framed it as a response to its concerns about the Illumina-Grail merger.[67] Guardant also claimed the lawsuit was filed in order to suppress competition in the marketplace.[68] |
訴訟 Czarnik 対イルミナ訴訟 2005年、共同創設者で元最高科学責任者のアンソニー・チャルニックがイルミナを提訴した。Czarnik v. Illumina Inc.事件では、裁判所はイルミナの訴えの一部を退ける申し立てを認めたが、発明者に関するチャルニックの主張は継続することを認めた。 コーネル大学とライフテクノロジーズによるイルミナに対する訴訟 2010年、コーネル大学とライフテクノロジーズはイルミナに対して訴訟を起こし、イルミナのマイクロアレイ製品が同大学が保有し、新興企業に独占的にラ イセンス供与された8件の特許を侵害していると主張した。この訴訟は2017年4月に過失の認定なしに和解した。2017年9月、両当事者は和解の見直し を求めた。コーネル大学はイルミナとライフテクノロジーズの両社に対して虚偽の陳述と詐欺を非難した。[56] コーネル大学は、サーモフィッシャーがイルミナとの訴訟の和解を約束し、コーネル大学で発明された他の特許に関する訴訟を提起できるように、マークマンの 文言を削除するよう求めたと主張した。サーモフィッシャーとイルミナは訴訟を提起する代わりに、カリフォルニア州で別の訴訟を解決し、極秘裏にそれらの まったく同じ特許をサブライセンスした。2018年、モニブ・ジルビ博士は、イルミナおよびその主要な従業員数名を相手取り、イルミナの特許申請に、バ ラーニ研究室で発明されたアイデアおよびZipCode DNA 配列が故意に組み込まれたと主張して、ニューヨーク南部地区で訴訟を起こした。この訴訟は却下されたが、イルミナおよびその弁護士が、これらの知的財産の 不正流用は「嵐の警告」であり、したがってこれらの特定の主張については時効が成立していると主張した後のことだった。また、モニブ・ジルビ博士は 2020年にニュージャージー州で、イルミナが創業初期に申請したNIHの主要助成金の非編集版コピーを要求する情報公開法に基づく訴訟を起こした。イル ミナの社内弁護士であるウィリアム・ヌーンは、2015年1月にこれら主要助成金の4件についても情報公開法に基づく請求を行っていた。 特許侵害訴訟 イルミナは、競合他社であるAriosa Diagnostics社に対する特許訴訟の当事者であった。この訴訟は、Verinata Health社がAriosa社を提訴した2012年に始まった。イルミナは2013年にベリナータを買収した後、訴訟に加わった。その後、アリオサはイ ルミナに対して反訴を起こした。[57] 裁判所はアリオサを支持する略式判決を下したが、連邦巡回区控訴裁判所はこれを覆した。[58] アリオサは当初、米国最高裁判所に上訴したが、裁判所が訴訟の受理を決定する前に両当事者は紛争を解決した。[59] 2016年2月、イルミナはオックスフォード・ナノポア・テクノロジーズを相手取って訴訟を起こした。イルミナは、オックスフォード・ナノポアが生物学的 ナノポアであるMycobacterium smegmatis porinA (MspA) のシーケンシングシステムへの利用に関する特許を侵害していると主張した。[60][61] 2016年8月、両者は訴訟で和解した。[62] 2020年2月、イルミナはBGIのシーケンシング製品「CoolMPS」に関連して特許侵害訴訟を起こした。[63] これに対してBGIは、連邦独占禁止法およびカリフォルニア州不正競争防止法違反で特許侵害訴訟を起こし、 特許を取得または行使し、BGIが米国市場に参入することを妨害したと主張している。[64] しかし、2022年5月、イルミナはDNAシーケンシングシステムの2件の特許を侵害したとして、カリフォルニア州のBGI米国法人に3億3300万ドル の支払いを命じられた。この訴訟の陪審は、イルミナが故意に特許を侵害したとし、BGIの侵害を主張した以前の主張は無効であるとも述べた。[65] 2022年5月6日、デラウェア地区連邦地方裁判所の陪審は、イルミナが故意にコンプリート・ゲノミクスが所有する2件の特許を侵害したとの評決を下し、 CGIに約3億3400万ドルの過去の損害賠償を認めた。陪審はまた、イルミナが所有する3件の特許を無効とした。[66] エルトゥーキーとタラサズに対する営業秘密訴訟 2022年3月、イルミナはガードアントの共同創業者であるヘルミー・エルトウキーとアミール・タラサズを営業秘密の窃盗で訴えた。[67] ガードアントは、この訴訟を「軽率で報復的なもの」と呼び、イルミナとグレイルの合併に対する懸念への対応であると主張した。[67] ガードアントはまた、この訴訟は市場での競争を抑制するために起こされたものであると主張した。[68] |
| https://en.wikipedia.org/wiki/Illumina,_Inc. |
リ ンク
文 献
そ の他の情報
Copyleft, CC, Mitzub'ixi Quq Chi'j, 1996-2099
☆
 ☆
☆